Mikrobielle Grenzflächenbiologie

 Im wissenschaftlichen Fokus der Forschungsgruppe „Mikrobielle Grenzflächenbiologie“ steht die Entwicklung innovativer, sowohl wirts- als auch erregergerichteter Therapieansätze zur Bekämpfung der Tuberkulose als globaler Gesundheitsbedrohung. Dabei konzentriert sich die FG auf zwei sich ergänzende Themenfelder: 1) Entschlüsselung von Wirtszellprozessen, die bei M. tuberculosis-Infektionen relevant sind, mit Schwerpunkt auf Makrophagen als zentrale zelluläre Nische, und 2) Identifizierung und Bewertung neuartiger Verbindungen mit antimykobakterieller Wirkung. Beide Themen werden mit einem breiten Spektrum zellbiologischer, biochemischer und mikrobiologischer Methoden bearbeitet.
Im wissenschaftlichen Fokus der Forschungsgruppe „Mikrobielle Grenzflächenbiologie“ steht die Entwicklung innovativer, sowohl wirts- als auch erregergerichteter Therapieansätze zur Bekämpfung der Tuberkulose als globaler Gesundheitsbedrohung. Dabei konzentriert sich die FG auf zwei sich ergänzende Themenfelder: 1) Entschlüsselung von Wirtszellprozessen, die bei M. tuberculosis-Infektionen relevant sind, mit Schwerpunkt auf Makrophagen als zentrale zelluläre Nische, und 2) Identifizierung und Bewertung neuartiger Verbindungen mit antimykobakterieller Wirkung. Beide Themen werden mit einem breiten Spektrum zellbiologischer, biochemischer und mikrobiologischer Methoden bearbeitet.
Durch umfassende Studien auf dem Gebiet der Wirt-Erreger-Interaktion haben wir in den letzten Jahren durch M. tuberculosis induzierte Signalwege identifiziert, die mit der Regulation des Lipidstoffwechsels in der Wirtszelle im Zusammenhang stehen. Daher streben wir derzeit die Entwicklung eines innovativen, wirtsgerichteten Therapieansatzes durch den Einsatz neuartiger, hochspezifischer niedermolekularer Inhibitoren an, die auf diese Signalwege abzielen und so den Zugang von M. tuberculosis zu Fettsäuren – seiner bevorzugten Kohlenstoffquelle in vivo – einschränken. Diese Studien umfassen den Einsatz neuartiger fluoreszierender Reporterstämme, Transkriptions-Knockdowns in M. tuberculosis sowie die Nutzung verschiedener zellulärer Modellsysteme. Ein besonderer Fokus liegt auf der Analyse der Biogenese und der Dynamik von Lipidtröpfchen sowie deren Interaktion mit dem Tuberkuloseerreger.
Darüber hinaus hat die FG ein langjähriges Interesse an der Identifikation und Charakterisierung neuer Verbindungen mit antimykobakterieller Wirkung. Die Gruppe ist integraler Bestandteil der präklinischen Wirkstoffpipeline sowohl am FZB als auch innerhalb der TTU Tuberkulose am Deutschen Zentrum für Infektionsforschung (DZIF). Die Expertise der Gruppe zeigt sich auch in ihrer aktiven Zusammenarbeit mit über 20 nationalen und internationalen Partnerinstitutionen in verschiedenen Projekten zur Erforschung von neuen TB-Wirkstoffen. Die Erweiterung der Kapazitäten und die Entwicklung neuer Methoden zur in vitro Wirkstoffprofilierung wird kontinuierlich vorangetrieben, einschließlich der laufenden Entwicklung genetisch veränderter M. tuberculosis-Stämme für eine effizientere Charakterisierung neuer Wirkstoffkandidaten und ihrer Zielstrukturen. Zusammen unterstreichen diese Ansätze unsere Position als wichtiger europäischer Partner in der in-vitro Arzneimittelprofilierung gegen Tuberkulose. Insgesamt verbindet die Gruppe erfolgreich grundlegende wissenschaftliche Fragen mit einer klaren translationalen Perspektive.
I. Der WNT-Signalweg in der M. tuberculosis-Infektion – von neu identifizierten Mediatoren zur wirtsgerichteten Therapie
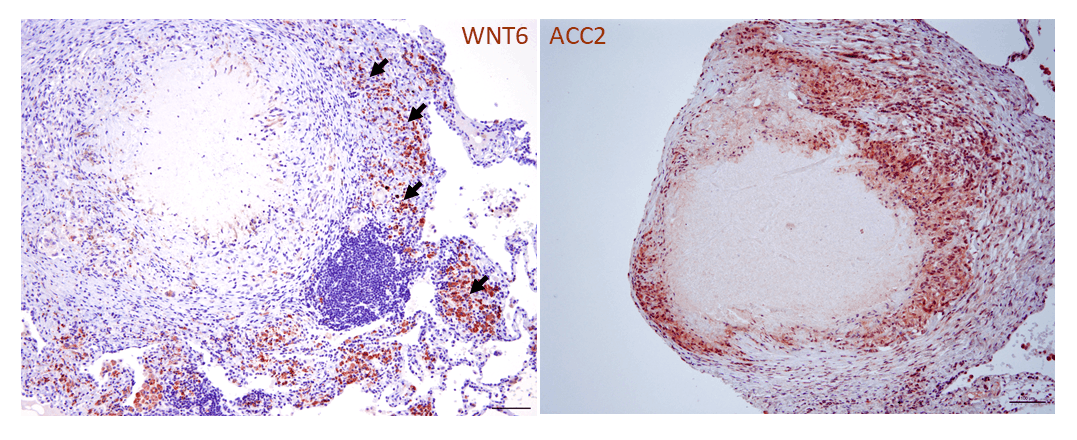
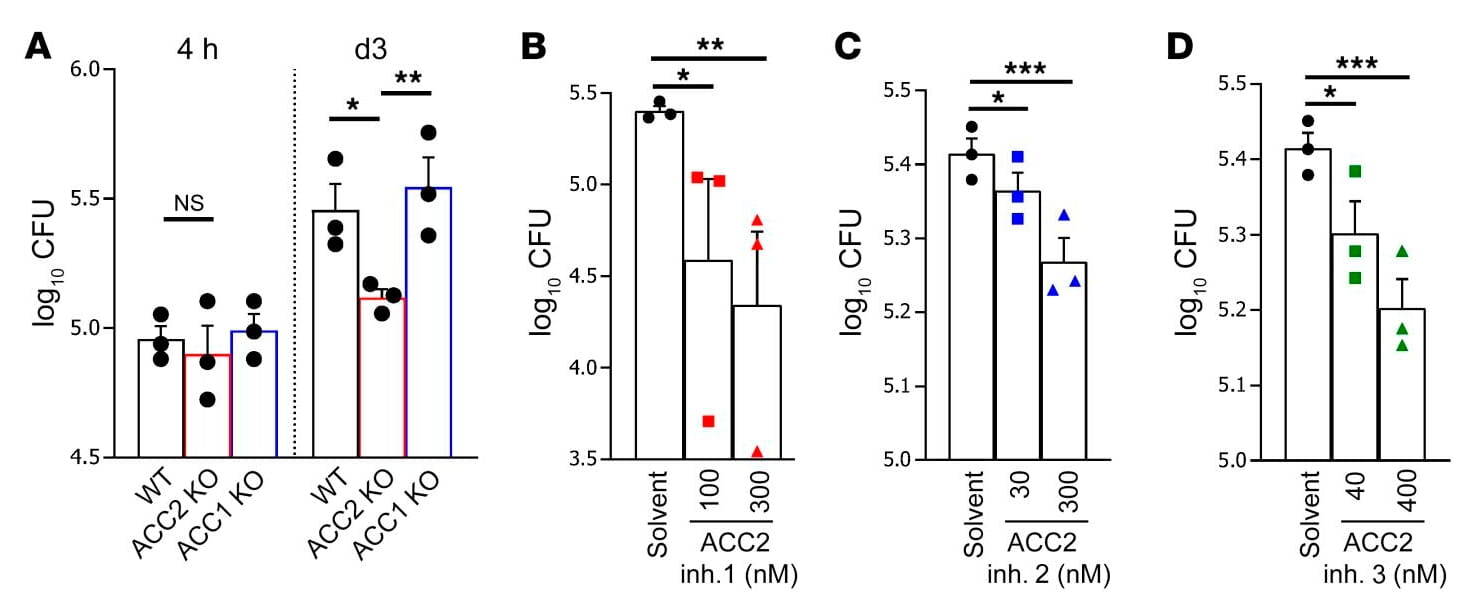
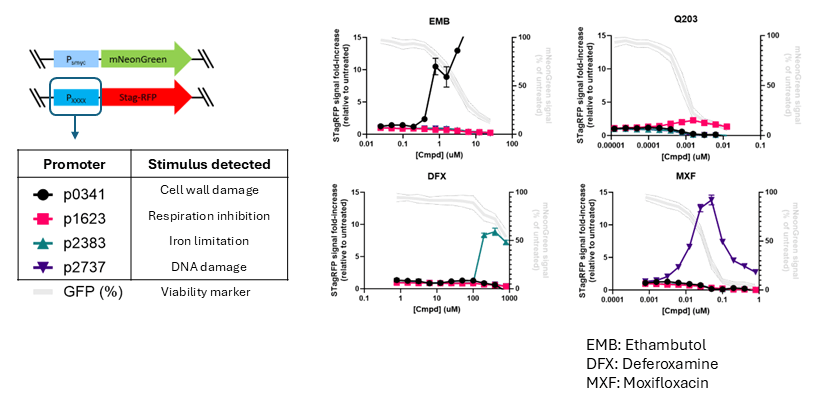
Aufbauend auf unseren früheren Arbeiten zur funktionellen Bedeutung von Muster-Erkennungs-Rezeptoren bei der Infektion mit M. tuberculosis konnten wir durch systematische Analysen mykobakterieninfizierter Makrophagen erstmals regulatorische Funktionen von Komponenten des evolutionär hochkonservierten Wingless/Integrase-1-(WNT-)Signalwegs in der pulmonalen Tuberkulose nachweisen. WNT-Proteine entfalten sowohl pro- als auch antiinflammatorische Effekte auf Makrophagen und andere Zellen des Immunsystems und können darüber hinaus die bakterielle Last während einer Infektion beeinflussen. In diesem Zusammenhang nimmt WNT6 eine besondere Rolle ein. Wir konnten eine ausgeprägte Expression von WNT6 und des durch WNT6 induzierten Enzyms Acetyl CoA-Carboxylase 2 (ACC2) sowohl in granulomatösen Läsionen der Lunge M. tuberculosis-infizierter Mäuse als auch im Lungengewebe von TB-Patientinnen und Patienten nachweisen (Abb. 1a; Brandenburg et al., J. Clin. Invest. 2021). Unter unserer Leitung und in enger Zusammenarbeit mit acht weiteren Forschungsgruppen am FZB konnte dann WNT6 durch umfassende funktionelle Analysen als ein zentraler Regulator der Bildung von Schaum-Makrophagen identifiziert werden. Diese lipidreichen Schaumzellen stellen während der Tuberkuloseinfektion ein zentrales intrazelluläres Habitat für M. tuberculosis dar. Durch die gezielte Modulation der WNT6-induzierten Signaltransduktion, insbesondere durch die spezifische genetische und pharmakologische Inhibition der Acetyl-CoA-Carboxylase 2 (Abb. 1b), gelang es uns, das Wachstum von M. tuberculosis sowohl in vitro als auch in vivo signifikant zu hemmen (Brandenburg et al., J. Clin. Invest. 2021). Die von uns identifizierte gezielte Beeinflussung des Neutrallipidstoffwechsels stellt damit einen vielversprechenden Ansatz für eine neue wirtsspezifische adjuvante Therapie der Tuberkulose dar (erteiltes Patent: Reiling N & Brandenburg J; EP3481392B1). Dieser Themenkomplex wird derzeit in weiterführenden Studien zum Lipidstoffwechsel während der M. tuberculosis-Infektion, unter anderem unter Verwendung von Mausstämmen mit unterschiedlicher Suszeptibilität, weiter vertieft.
Ausgewählte Publikationen zum Projekt:
- Brandenburg J, et al. J Clin Invest. 2021 Aug 16;131(16):e141833. doi: 10.1172/JCI141833.
- Reiling N & Brandenburg J; EP3481392B1 (patent granted).
- Brandenburg J, et al.; ACS Infect Dis. 2022 Jul 8;8(7):1303-1315. doi: 10.1021/acsinfecdis.2c00075.
- Brandenburg J & Reiling N. Front Immunol. 2016 Dec 26;7:635. doi: 10.3389/fimmu.2016.00635.
- Schaale K, et al.; J Immunol. 2013 Nov 15;191(10):5182-95. doi: 10.4049/jimmunol.1201819.
- Schaale K, et al. Eur J Cell Biol. 2011 Jun-Jul;90(6-7):553-9. doi: 10.1016/j.ejcb.2010.11.004.
- Neumann J, et al.; FASEB J. 2010 Nov;24(11):4599-612. doi: 10.1096/fj.10-160994.
- Blumenthal A, et al.; Blood. 2006 Aug 1;108(3):965-73. doi: 10.1182/blood-2005-12-5046.
II. Tuberkulostearinsäure-haltige Phosphatidylinositole als Marker für die bakterielle Belastung bei Tuberkulose
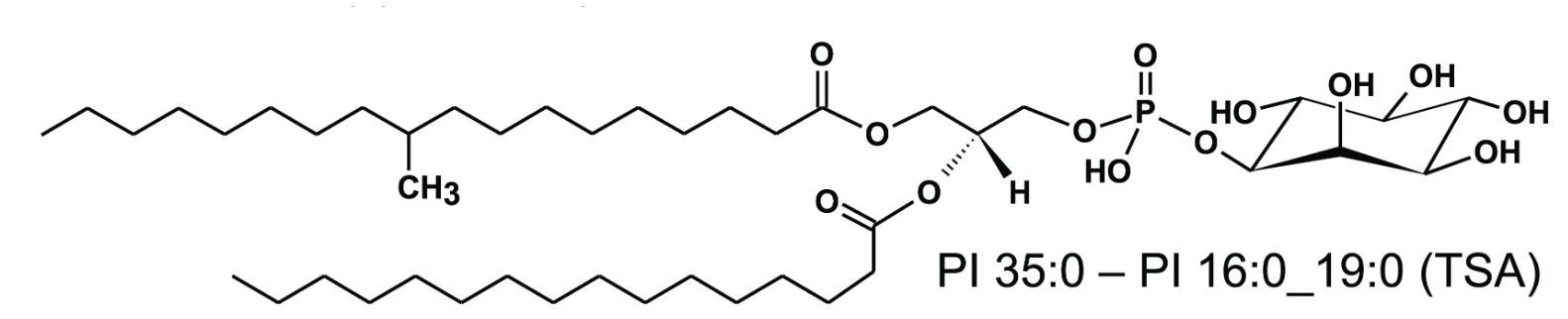
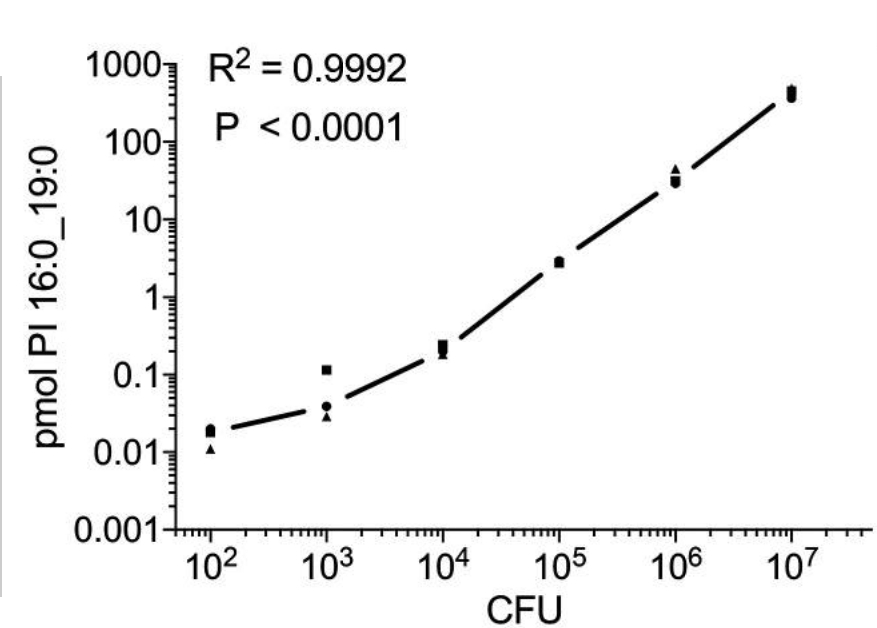
Von translationaler Bedeutung – hier jedoch mit Blick auf die Diagnostik der Tuberkulose – ist zudem der Nachweis spezifischer M. tuberculosis-Lipide, die als Biomarker zur Überwachung der Tuberkulosetherapie eingesetzt werden können. Mithilfe lipidomischer Ansätze und in enger Zusammenarbeit mit der Forschungsgruppe Bioanalytische Chemie konnten wir zeigen, dass Phosphatidylinositole (PI), die Tuberkulostearinsäure (TSA) enthalten, molekulare Marker für eine Infektion mit klinisch relevanten Stämmen des M. tuberculosis-Komplexes darstellen und die bakterielle Last widerspiegeln (Brandenburg et al., ACS Infect. Dis. 2022). Für den am häufigsten vorkommenden Lipidmarker wurden Nachweisgrenzen von ca. 10² bzw. 10³ koloniebildenden Einheiten (CFU) für bakterielle bzw. Zellkultursysteme bestimmt (Abb. 2). Es wurde eine vollständige analytische Methodik einschließlich Probenaufarbeitung entwickelt, die innerhalb eines Tages durchgeführt werden kann und damit etwa 30-mal schneller ist als konventionelle, kultur-basierte Verfahren. Mithilfe dieser indirekten und kulturfreien Nachweismethode konnten wir die Erregerlast in infizierten murinen Makrophagen, humanen Neutrophilen sowie in murinem Lungengewebe bestimmen. Diese aus mykobakteriellen PIs abgeleiteten Markerlipide wurden in höheren Konzentrationen in mononukleären Zellen des peripheren Blutes von TB-PatientInnen nachgewiesen als bei gesunden Kontrollpersonen. Darüber hinaus zeigten sich in einer kleinen Kohorte antibiotika-responsiver TB-PatientInnen zu Therapiebeginn erhöhte Konzentrationen dieser molekularen Marker, die nach erfolgreicher anti-tuberkulöser Behandlung abnahmen. Somit kann die Konzentration von TSA-haltigen Phosphatidylinositolen als Korrelat der mykobakteriellen Last in experimentellen Modellen und in-vitro Systemen dienen und stellt potenziell auch ein klinisch relevantes Instrument zur Überwachung der Krankheitsaktivität bei TB-PatientInnen dar.
Ausgewählte Publikationen zum Projekt:
- Brandenburg J, et al. J Clin Invest. 2021 Aug 16;131(16):e141833. doi: 10.1172/JCI141833.
- Brandenburg J, et al.; ACS Infect Dis. 2022 Jul 8;8(7):1303-1315. doi: 10.1021/acsinfecdis.2c00075.
III. Multiple Strategien von M. tuberculosis zur Überlebenssicherung unter Spermin induziertem Stress
Die Entdeckung von Molekülen, die die Behandlungsdauer der Tuberkulose (TB) verkürzen können, birgt das Potenzial, die derzeitigen Therapieschemata deutlich zu verbessern. Polyamine sind organische, polykationische Alkylamine, die in millimolaren Konzentrationen in allen lebenden Zellen vorkommen. Spermin (Spm) ist ein Polyamin, das bereits vor über 70 Jahren als toxisch für M. tuberculosis beschrieben wurde. Streptomyces coelicolor, ein naher Verwandter von M. tuberculosis, nutzt einen γ-Glutamylierungsweg, um Spermin funktionell zu neutralisieren. Vor diesem Hintergrund untersuchten wir, ob ein ähnlicher Mechanismus auch in M. tuberculosis aktiv ist und wie sich dieses Bakterium gegen die toxischen Effekte von Spermin schützt. Wir konnten zeigen, dass Spermin das Wachstum von M. tuberculosis hemmt und dass das Enzym GlnA3Mt (Rv1878) als γ-Glutamylspermin-Synthetase fungiert, wobei es bevorzugt Spermin gegenüber anderen Polyaminen umsetzt. Allerdings führte die Deletion des glnA3-Gens nicht zu einer erhöhten Empfindlichkeit gegenüber Spermin. Auch wurde die Expression des Gens durch Spermin nicht beeinflusst. Stattdessen wurde das Effluxpumpen-Gen mmr (Rv3065) stark hochreguliert, was darauf hindeutet, dass die Entgiftung von Spermin möglicherweise primär durch aktiven Export und nicht durch enzymatische Modifikation erfolgen könnte. Weitere Experimente zeigten unterschiedliche Rollen weiterer Effluxpumpen und Enzyme in der Spermin-Resistenz und anderen Antworten auf chemischen Stress. Die Δrv1877-Mutante war empfindlich gegenüber Spermin-induziertem Stress, was auf eine Beteiligung von Rv1877 (einer multidrug-MFS-Typ-Effluxpumpe) am Export von Spermin hindeutet. Im Gegensatz dazu war die Δrv0191-Mutante unempfindlich gegenüber Spermin, zeigte jedoch eine erhöhte Sensitivität gegenüber oxidativem Stress, was auch auf eine Rolle beim Transport niedermolekularer Thiolverbindungen schließen liesse. Die Δrv1878-Mutante (GlnA3-defizient) zeigte ein verstärktes Wachstum unter Eisenmangel, war jedoch empfindlicher gegenüber Zellwandstress, was darauf hindeutet, dass Rv1878 zur Eisenhomöostase und zur Aufrechterhaltung der Zellwandintegrität beitragen könnte. Insgesamt legen unsere Studien nahe, dass M. tuberculosis mehrere molekulare Mechanismen einsetzt, um die Toxizität von Spermin sowie die damit verbundene zelluläre Stresssituation zu überleben.
Ausgewählte Publikationen zum Projekt:
- Sao Emani C & Reiling N; Microbiol Spectr. 2024 Jan 11;12(1):e0356823. doi: 10.1128/spectrum.03568-23.
- Sao Emani C & Reiling N; Front Microbiol. 2024 Mar 4;15:1359188. doi: 0.3389/fmicb.2024.1359188.
- Krysenko S, et al. J Bacteriol. 2025 Feb 20;207(2):e0043924. doi: 10.1128/jb.00439-24.
IV. "New drugs wanted" - Schnelle und relevante Testsysteme zur Identifizierung neuer Wirkstoffe gegen M. tuberculosis
Im Jahr 2024 hat die Tuberkulose erneut ihre Position als häufigste Todesursache durch einen einzelnen Infektionserreger eingenommen und zählt derzeit zu den zehn häufigsten Todesursachen weltweit. Daraus ergibt sich ein dringender Bedarf an neuen Antibiotika zur Verbesserung der Behandlungsergebnisse von Tuberkulosepatientinnen und -patienten, insbesondere bei Infektionen mit Stämmen, die gegen die derzeit besten verfügbaren Erstlinienmedikamente Isoniazid und Rifampicin resistent sind. Vor diesem Hintergrund haben wir in den vergangenen Jahren verschiedene in-vitro Testsysteme etabliert, um potenzielle neue Anti-TB-Wirkstoffe schnell und zuverlässig zu identifizieren und zu charakterisieren. Von fluoreszenzbasierten Hochdurchsatz-Screenings von Substanzbibliotheken im 384-Well-Format und dosisabhängigen Assays zur Wachstumshemmung (IC₅₀-Bestimmung) über Time-Kill-Assays, Aktivitätstests in Makrophagen, die Generierung resistenter Mutanten bis hin zu maßgeschneiderten Methoden zur Zielstrukturidentifikation und -validierung spannt unsere umfassende Pipeline zur frühen Identifizierung und Charakterisierung antituberkulöser Wirkstoffe eine entscheidende Brücke zwischen Hit-Identifizierung und Lead-Entwicklung innerhalb der deutschen und europäischen TB-Wirkstoffentwicklungslandschaft.
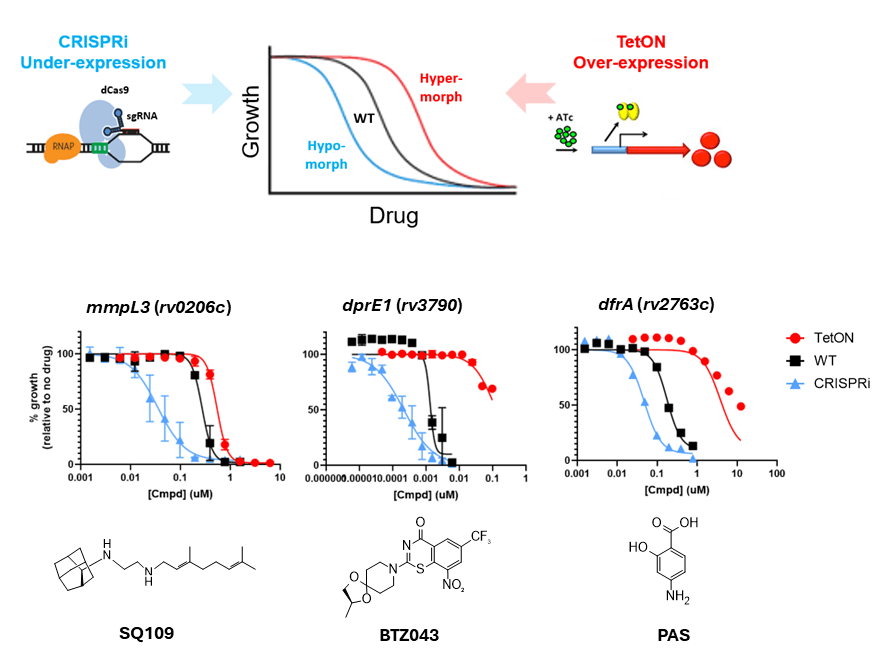
Ein zentraler Schwerpunkt unserer Arbeiten in der Wirkstoffentwicklung liegt auf der Entwicklung und Implementierung von Werkzeugen zur Beschleunigung der Aufklärung von Wirkmechanismus (Mode of Action, MoA) und molekularer Zielstruktur – ein Prozess, der in frühen Phasen der phänotypischen Wirkstoffforschung häufig einen Engpass darstellt. Die Bestimmung des Wirkmechanismus eines Wirkstoffs kann sowohl die spätere Lead-Optimierung, etwa durch strukturgeleitetes Wirkstoffdesign, erleichtern als auch dazu beitragen, Substanzen mit unerwünschten Zielstrukturen möglichst frühzeitig zu depriorisieren und damit Ressourcen für vielversprechende Kandidaten zu schonen. In diesem Zusammenhang nutzen wir gezielt die Möglichkeiten genetischer Modifikationen, um Panels rekombinanter M. tuberculosis-Stämme zu generieren, die die Identifizierung von Wirkmechanismen und Zielstrukturen erheblich beschleunigen.
In einem Ansatz erzeugen wir fluoreszierende „Reporter-Stämme“: Dies ermöglichen durch eine transkriptionelle Fusion eines stimulus-spezifisch responsiven Promotors mit einem Fluoreszenzproteingen eine schnelle Bewertung (von 2–3 Tagen) spezifischer, durch neue Wirkstoffkandidaten induzierter Stressantworten im Tuberkuloseerreger (siehe Abb. 3). In einem zweiten Ansatz nutzen wir die gezielte Hoch- und Herunterregulation einzelner Gene zur Analyse der Wirkstoff-Zielstruktur-Interaktion auf Zellebene, basierend auf dem Konzept der differenziellen Wirkstoffsensitivität infolge unterschiedlicher intrazellulärer Zielprotein-Konzentrationen (siehe Abb. 4). In beiden Fällen erweitern wir unsere Stammsammlungen kontinuierlich, um ein möglichst breites Spektrum an Wirkmechanismen und Zielstrukturen untersuchen zu können. Sollten die hier beschriebenen Werkzeuge oder Techniken für Ihre eigene Forschung von Interesse sein oder Sie verfügen über Substanzen, die Sie in Ihrer Wirksamkeit gegen M. tuberculosis charakterisieren möchten, zögern Sie bitte nicht, uns zu kontaktieren. Wir sind stets sehr interessiert daran, neue Kooperationen in der Wirkstoffforschung einzugehen.
Wir sind sowohl an Zielstruktur-gerichteten als auch an phänotypischen Screenings mit verschiedenen Kooperationspartnern beteiligt und verfügen über enge Verbindungen zu zahlreichen medizinisch-chemischen und organisch-chemischen Arbeitsgruppen in Europa und darüber hinaus, über die wir Zugang zu entscheidendem Ausgangsmaterial für die Identifizierung neuer antituberkulöser Wirkstoffe erhalten. Wir sind stolz darauf, Teil der Thematischen Translationalen Transfer Unit Tuberculosis (TTU-TB) des Deutschen Zentrums für Infektionsforschung (DZIF) zu sein, die sich mit dem Themenfeld „Neue Wirkstoffe und Therapieschemata“ befasst. Darüber hinaus waren wir Partner der EU-geförderten Marie-Skłodowska-Curie-Maßnahme „MepAnti“ (einem Ausbildungsnetzwerk zur Entwicklung neuer Antiinfektiva) sowie in mehreren vom BMBF/BMFTR geförderten Forschungsverbünden. Kürzlich haben wir zudem ein High-Content-Imaging-System etabliert, das es uns ermöglicht, auch Wirkstoffe zu identifizieren, die nicht den Erreger selbst, sondern den Wirt adressieren, mit dem Ziel, aktive Substanzen für zukünftige wirtsgerichtete Therapieansätze zu identifizieren.
Ausgewählte Publikationen zum Projekt:
- Kiefer AF, et al.; JACS Au. 2025 Nov 18;5(12):6060-6071. doi: 10.1021/jacsau.5c00950.
- Palme PR, et al.; J Med Chem. 2025 Dec 11;68(23):25274-25289. doi: 10.1021/acs.jmedchem.5c02284.
- Braun-Cornejo M, et al.; JACS Au. 2025 Feb 21;5(3):1146-1156. doi: 10.1021/jacsau.4c00935.
- Braun-Cornejo M, et al.; ACS Omega. 2024 Aug 29;9(36):38160-38168. doi: 10.1021/acsomega.4c05537.
- Du X, et al.; ChemMedChem. 2023 Sep 1;18(17):e202300279. doi: 10.1002/cmdc.202300279.
- Johannsen S, et al.; ChemMedChem. 2023 Jun 1;18(11):e202200590. doi: 10.1002/cmdc.202200590.
- Zhu D, et al.; Chem Sci. 2022 Aug 8;13(36):10686-10698. doi: 10.1039/d2sc02371g.
- Richter A, et al.; ACS Med Chem Lett. 2022 Jul 25;13(8):1302-1310. doi: 10.1021/acsmedchemlett.2c00215. Aryal N, et al.; J Nat Prod. 2022 Mar 25;85(3):530-539. doi: 10.1021/acs.jnatprod.1c01051.
- Brandenburg J, et al. ; J Clin Invest. 2021 Aug 16;131(16):e141833.
- Jumde RP, et al.; Chem Sci. 2021 Apr 28;12(22):7775-7785. doi: 10.1039/d1sc00330e.
- “New drugs & regimens: drug screening and improving the predictive value of preclinical models” – TTU TB, Deutsches Zentrum für Infektionsforschung (DZIF), BMBF, 2021-2025
- “Validation of the confirmed γ-glutamylspermine synthetase GlnA3 as a target for development of novel anti-tubercular drugs” - Sub project: "Relevance of the γ-glutamylspermine synthetase GlnA3 for successful growth inhibition of M. tuberculosis" (GSS-TUBTAR) – BMBF, Programm: Targetvalidierung für die pharmazeutische Wirkstoffentwicklung II, 2021-2023
- “Isolation and Characterization of M. tuberculosis-induced lipid droplets from human primary macrophages“; Leibniz Center Infection (LCI), 2018-2021
- “Discovering new therapeutic targets and drugs to combat AMR tuberculosis: proteomics characterization and drug screening of mycobacterium-infected macrophages" – Olav Thon Foundation, Norwegen (in Kooperation with T. Flo (N), M. Lerm (SE) und K. Prasad (IND), 2018-2022
- „Exploiting the methylerythritol phosphate pathway as a source of drug targets for novel anti-infectives“ - MepAnti - Marie Skodowska-Curie Innovative Training Networks, 2020-2024
- „NaPAnti: Development of natural product-based antibacterials targeting the sliding clamp DnaN“ - Bundesministerium für Bildung und Forschung, 2019-2022
- „InhibitMycoRex: Optimization of mycobacterial thioredoxin reductase inhibitors, novel lead compounds against M. tuberculosis“ – Deutsches Zentrum für Infektionsforschung, 2019-2021
- „ETBRA: European tuberculosis regimen accelerator“ – EU / Horizon 2020 (IMI), 2020-
- Isolierung und Kultivierung von primären Immunzellen
- murine Knochenmarkmakrophagen
- humane Monozyten
- aus Blutmonozyten differenzierte Makrophagen
- humane Alveolarmakrophagen
- In vitro-Infektion von Primärzellen mit M. tuberculosis (Mtb) unter L3 und S3-Bedingungen
- Magnetische Isolierung von Mtb-enthaltenden intrazellulären Kompartimenten
- Magnetische Isolierung und Charakterisierung von Makropinosomen
- Isolierung und funktionelle Charakterisierung von Lipidkörpern
- CRISPR/CAS9 in humanen Immunzellen
- Transfektion von humanen Makrophagen
- Quantitative RT-PCR (Light Cycler)
- ELISA
- Histologie und Immunhistologie
- Durchflusszytometrie von Mykobakterien und Mtb-infizierten Immunzellen (BSL3)
- Aerosol-Infektion von immunkompetenten und selektiv defizienten Mäusen mit Mtb
- Identifikation of neuer Substanzen mit antimycobacterieller Aktivität
- 96-Well- Format basierte anti-Tb Aktivitätstests mit GFP- und mCherry-exprimierenden Mtb Bakterien (H37Rv)
- Echtzeit- Makrophagen-Zytotoxizitätstests (xCelligence)
- Aktivitätstests mit Mtb-infizierten humanen Makrophagen
- Aktivitätstests mit suszeptiblen, MDR und XDR-Mtb Stämmen (in Koop. mit Dr. Doris Hillemann, Nationales Referenz Zentrum) (MGIT Format)
2026
Tulu, B, Brehm, TT, van Crevel, R, Dallenga, T, DiNardo, AR, Dheda, K, Eggeling, J, Enbiale, W, Gröschel, MI, Hao, J, Kumar, V, van Laarhoven, A, Londt, R, Prosser, G, Randall, P, Reiling, N, Rybniker, J, Schaible, UE, Schurr, E, Suarez, I, Theobald, SJ, Wilkinson, RJ & Lange, C 2026, 'Host- and pathogen-related determinants of pulmonary versus extrapulmonary tuberculosis', European Respiratory Review, Jg. 35, Nr. 179. https://doi.org/10.1183/16000617.0174-2025
Walsh, DJ, Hamid, R, Giele, T, Reiling, N, Rottmann, M, Hamed, MM & Hirsch, AKH 2026, 'Design and optimization of simplified inhibitors targeting Escherichia coli and Klebsiella pneumoniae IspE', RSC medicinal chemistry. https://doi.org/10.1039/d5md00874c
Yazgili, AS, Giotopoulou, GA, Behrend, SJ, Koops, F, Neiens, V, Meul, T, Zemke, L, Reiling, N, Goldmann, T, Stathopoulos, GT & Meiners, S 2026, 'PA200 differentially regulates the proteasome and inhibits migration of NSCLC cells', JOURNAL OF CELL SCIENCE , Jg. 139, Nr. 4. https://doi.org/10.1242/jcs.264683
2025
Abdelaziz, R, Dube, M, Mann, L, Richter, A, Robaa, D, Reiling, N, Abdel-Halim, M & Imming, P 2025, 'Synthesis and Antimycobacterial Assays of Some New Ethambutol Analogs', Molecules (Basel, Switzerland), Jg. 30, Nr. 3. https://doi.org/10.3390/molecules30030600
Braun-Cornejo, M, Platteschorre, M, de Vries, V, Bravo, P, Sonawane, V, Hamed, MM, Haupenthal, J, Reiling, N, Rottmann, M, Piet, D, Maas, P, Diamanti, E & Hirsch, AKH 2025, 'Positive Charge in an Antimalarial Compound Unlocks Broad-Spectrum Antibacterial Activity', JACS Au, Jg. 5, Nr. 3, S. 1146-1156. https://doi.org/10.1021/jacsau.4c00935
Kiefer, AF, Voltz, A, Scherzer, D, Reberšek, R, Kany, AM, Prosser, G, Neuber, M, Reiling, N, Hirsch, AKH & Müller, R 2025, 'Engineering an Artificial Myxopyronin Derivative with Enhanced Metabolic Stability via Mutasynthesis', JACS Au. https://doi.org/10.1021/jacsau.5c00950
Krysenko, S, Emani, CS, Bäuerle, M, Oswald, M, Kulik, A, Meyners, C, Hillemann, D, Merker, M, Prosser, G, Wohlers, I, Hausch, F, Brötz-Oesterhelt, H, Mitulski, A, Reiling, N & Wohlleben, W 2025, 'GlnA3Mt is able to glutamylate spermine but it is not essential for the detoxification of spermine in Mycobacterium tuberculosis', JOURNAL OF BACTERIOLOGY, S. e0043924. https://doi.org/10.1128/jb.00439-24
Nitschkowski, D, Vierbuchen, T, Heine, H, Behrends, J, Reiling, N, Reck, M, Rabe, KF, Kugler, C, Ammerpohl, O, Drömann, D, Muley, T, Kriegsmann, M, Stathopoulos, GT, Arendt, KAM, Goldmann, T & Marwitz, S 2025, 'SMAD2 linker phosphorylation impacts overall survival, proliferation, TGFβ1-dependent gene expression and pluripotency-related proteins in NSCLC', BRITISH JOURNAL OF CANCER . https://doi.org/10.1038/s41416-025-02970-1
Palme, PR, Grover, S, Abdelaziz, R, Mann, L, Kany, AM, Ouologuem, L, Bartel, K, Sonnenkalb, L, Reiling, N, Hirsch, AKH, Schnappinger, D, Rubinstein, JL, Imming, P & Richter, A 2025, 'Design, Synthesis, and Biological Evaluation of Mono- and Diamino-Substituted Squaramide Derivatives as Potent Inhibitors of Mycobacterial Adenosine Triphosphate (ATP) Synthase', JOURNAL OF MEDICINAL CHEMISTRY. https://doi.org/10.1021/acs.jmedchem.5c02284
Riegler-Berket, L, Gödl, L, Polidori, N, Aschauer, P, Grininger, C, Prosser, G, Lichtenegger, J, Sagmeister, T, Parigger, L, Gruber, CC, Reiling, N & Oberer, M 2025, 'M. tuberculosis meets European lead factory – Identification and structural characterization of novel Rv0183 inhibitors using X-ray crystallography', Diseases and Therapeutics, Jg. 1. https://doi.org/10.1016/j.dist.2025.100002
2024
Braun-Cornejo, M, Ornago, C, Sonawane, V, Haupenthal, J, Kany, AM, Diamanti, E, Jézéquel, G, Reiling, N, Blankenfeldt, W & Maas, P et al. 2024, 'Target-Directed Dynamic Combinatorial Chemistry Affords Binders of Mycobacterium tuberculosis IspE', ACS omega, Jg. 9, Nr. 36, S. 38160-38168. https://doi.org/10.1021/acsomega.4c05537
Kotimoole, CN, Ramya, VK, Kaur, P, Reiling, N, Shandil, RK, Narayanan, S, Flo, TH & Prasad, TSK 2024, 'Discovery of Species-Specific Proteotypic Peptides To Establish a Spectral Library Platform for Identification of Nontuberculosis Mycobacteria from Mass Spectrometry-Based Proteomics', JOURNAL OF PROTEOME RESEARCH , Jg. 23, Nr. 3, S. 1102-1117. https://doi.org/10.1021/acs.jproteome.3c00850
Sao Emani, C & Reiling, N 2024, 'The efflux pumps Rv1877 and Rv0191 play differential roles in the protection of Mycobacterium tuberculosis against chemical stress', Frontiers in Microbiology, Jg. 15, S. 1359188. https://doi.org/10.3389/fmicb.2024.1359188
2023
Du, X, Sonawane, V, Zhang, B, Wang, C, de Ruijter, B, Dömling, ASS, Reiling, N & Groves, MR 2023, 'Inhibitors of Aspartate Transcarbamoylase inhibit Mycobacterium tuberculosis growth', ChemMedChem, S. e202300279. https://doi.org/10.1002/cmdc.202300279
Johannsen, S, Gierse, RM, Olshanova, A, Smerznak, E, Laggner, C, Eschweiler, L, Adeli, Z, Hamid, R, Alhayek, A, Reiling, N, Haupenthal, J & Hirsch, AKH 2023, 'Not Every Hit-Identification Technique Works on 1-Deoxy-d-Xylulose 5-Phosphate Synthase (DXPS): Making the Most of a Virtual Screening Campaign', ChemMedChem, Jg. 18, Nr. 11, S. e202200590. https://doi.org/10.1002/cmdc.202200590
Kotimoole, C, Antil, N, Kasaragod, S, Behera, S, Arvind, A, Reiling, N, Flo, T & Prasad, T 2023, 'Development of a spectral library for the discovery of altered genomic events in Mycobacterium avium associated with virulence using mass spectrometry-based proteogenomic analysis', MOLECULAR & CELLULAR PROTEOMICS, Jg. 22, Nr. 5, S. 100533. https://doi.org/10.1016/j.mcpro.2023.100533
Seitz, L, Reiling, N & Hilgeroth, A 2023, 'Synthesis and Evaluation of Novel Substituted N-Aryl 1,4-Dihydropyridines as An-tituberculostatic Agents', Medicinal Chemistry. https://doi.org/10.2174/1573406419666230622121512
2022
Aryal N, Chen J, Bhattarai K, Hennrich O, Handayani I, Kramer M, Straetener J, Wommer T, Berscheid A, Peter S, Reiling N, Brötz-Oesterhelt H, Geibel C, Lämmerhofer M, Mast Y, Gross H. High Plasticity of the Amicetin Biosynthetic Pathway in Streptomyces sp. SHP 22-7 Led to the Discovery of Streptcytosine P and Cytosaminomycins F and G and Facilitated the Production of 12F-Plicacetin. J NAT PROD. 2022 Mar 25;85(3):530-539. doi: 10.1021/acs.jnatprod.1c01051. Epub 2022 Mar 9.
Brandenburg J, Heyckendorf J, Marwitz F, Zehethofer N, Linnemann L, Gisch N, Karaköse H, Reimann M, Kranzer K, Kalsdorf B, Sanchez-Carballo P, Weinkauf M, Scholz V, Malm S, Homolka S, Gaede KI, Herzmann C, Schaible UE, Hölscher C, Reiling N, Schwudke D. Tuberculostearic Acid-Containing Phosphatidylinositols as Markers of Bacterial Burden in Tuberculosis. ACS INFECT DIS. 2022 Jul 8;8(7):1303-1315. doi: 10.1021/acsinfecdis.2c00075.
Gisch N, Utpatel C, Gronbach LM, Kohl TA, Schombel U, Malm S, Dobos KM, Hesser DC, Diel R, Götsch U, Gerdes S, Shuaib YA, Ntinginya NE, Khosa C, Viegas S, Kerubo G, Ali S, Al-Hajoj SA, Ndung'u PW, Rachow A, Hoelscher M, Maurer FP, Schwudke D, Niemann S, Reiling N, Homolka S. Sub-Lineage Specific Phenolic Glycolipid Patterns in the Mycobacterium tuberculosis Complex Lineage 1. FRONT MICROBIOL. 2022 Mar 8;13:832054. doi: 10.3389/fmicb.2022.832054.
Hansen J, Kolbe K, König IR, Scherließ R, Hellfritzsch M, Malm S, Müller-Loennies S, Zallet J, Hillemann D, Wiesmüller KH, Herzmann C, Brandenburg J, Reiling N. Lipobiotin-capture magnetic bead assay for isolation, enrichment and detection of Mycobacterium tuberculosis from saliva. PLOS ONE. 2022 Jul 15;17(7):e0265554. doi: 10.1371/journal.pone.0265554.
Richter A, Seidel RW, Goddard R, Eckhardt T, Lehmann C, Dörner J, Siersleben F, Sondermann T, Mann L, Patzer M, Jäger C, Reiling N, Imming P. BTZ-Derived Benzisothiazolinones with In Vitro Activity against Mycobacterium tuberculosis. ACS MED. CHEM. LETT. 2022, July 25, 2022 epub ahead of print. https://doi.org/10.1021/acsmedchemlett.2c00215
Zhu D, Johannsen S, Masini T, Simonin C, Haupenthal J, Illarionov B, Andreas A, Awale M, Gierse RM, van der Laan T, van der Vlag R, Nasti R, Poizat M, Buhler E, Reiling N, Müller R, Fischer M, Reymond JL & Hirsch A. Discovery of novel drug-like antitubercular hits targeting the MEP pathway enzyme DXPS by strategic application of ligand-based virtual screening. CHEM SCI. 2022, doi: 10.1039/D2SC02371G Epub 2022 Aug 8.
Leitung der Forschungsgruppe

Wissenschaftliche Mitarbeiter:innen


Technische Mitarbeiter:innen