Wirts-Mikroben Interaktom

Tuberkulose (TB), verursacht durch das Bakterium Mycobacterium tuberculosis, gehört nach wie vor zu den tödlichsten Infektionskrankheiten weltweit. Millionen Menschen sind jedes Jahr betroffen, und das zunehmende Auftreten multiresistenter und persistierender Infektionen unterstreicht den dringenden Bedarf an innovativer Forschung an Wirts-Pathogen-Interaktionen. Unsere Forschungsgruppe untersucht auf zellulärer und molekularer Ebene, wie pathogene Mykobakterien den Lipidstoffwechsel der Wirtszelle ausnutzen, um zu überleben, zu persistieren und Krankheiten auszulösen.
Das Labor der FB Barisch befindet sich am CSSB in Hamburg (https://www.cssb-hamburg.de/) und Teils des FZBs als auch an den Fachbereich Biologie der Universität Hamburg angeschlossen. Diese einzigartige Konstellation ermöglicht es uns, modernste bildgebende Verfahren, massenspektrometrische Lipidomics und Infektionsbiologie in einem multidisziplinären Umfeld zu kombinieren.
Forschungsschwerpunkte
Ziel der Gruppe ist es, zu verstehen, wie intrazelluläre Mykobakterien auf Wirtslipide – wie Fettsäuren, Sterole und komplexe Lipide – zugreifen, um:
- Energie zu gewinnen: durch Katabolisierung von Wirtslipiden und deren Integration in den bakteriellen Stoffwechsel.
- Ihre charakteristische, lipidreiche Zellhülle aufzubauen: Mykobakterien besitzen eine komplexe, stark lipidreiche Zellwand, die entscheidend für Virulenz und Persistenz ist.
- Speicherlipide während der Infektion zu produzieren: was ihre langfristige intrazelluläre Überlebensfähigkeit unterstützt.
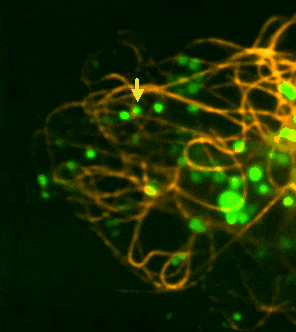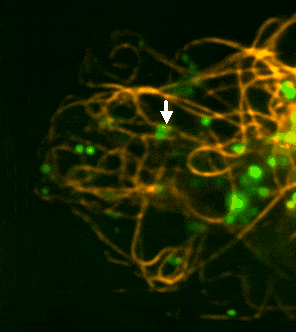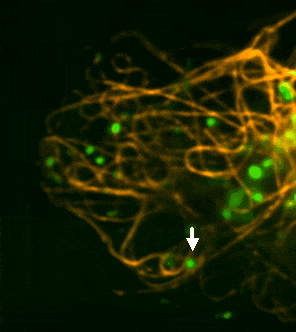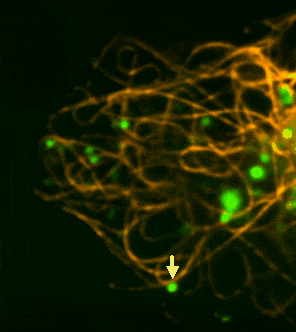
Während der Infektion induziert der Erreger aktiv die Bildung „schaumiger“ Makrophagen, die zahlreiche Lipidtröpfchen (LDs) enthalten – ideale Nischen für Nährstoffversorgung und Überleben. Mithilfe des Modells Dictyostelium discoideum infiziert mit Mycobacterium marinum konnten wir zeigen, dass Mykobakterien bei fehlenden LDs verstärkt auf ER-abgeleitete Phospholipide als Nährstoffquelle zurückgreifen.
Unsere Forschung basiert auf drei wissenschaftlichen Säulen: Mykobakterien, Mikroskopie und Lipide.


Unser Ansatz
Wir untersuchen mikrobielle-Wirts-Lipidinteraktionen durch Kombination von:
- Modellsystemen wie Dictyostelium discoideum infiziert mit Mycobacterium marinum, um mechanistische Einblicke zu gewinnen.
- Fortgeschrittener Mikroskopie (Live-Cell, Spinning Disc, Lattice Light Sheet), korrelativer Licht-Elektronenmikroskopie ((cryo)CLEM) sowie Hochdruckgefrieren/Tomographie.
- Funktionalisierten Lipidsonden, massenspektrometrischen Lipidomics und quantitativer Verfolgung von Lipidflüssen zwischen Wirt und Pathogen.
- Humanen Infektionsmodellen (genetisch modifizierte Makrophagen), um Erkenntnisse in medizinisch relevante Systeme zu übertragen.
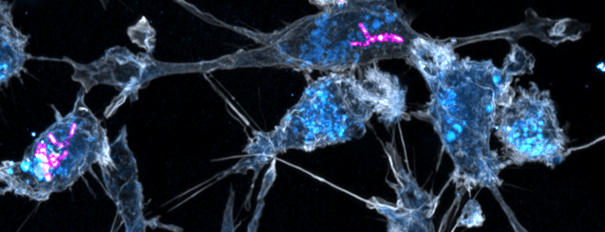 M. marinum-infizierte humane Makrophagen. Aktin: grau, Endosomen: blau, M. marinum: magenta.
M. marinum-infizierte humane Makrophagen. Aktin: grau, Endosomen: blau, M. marinum: magenta.
Warum das wichtig ist
Indem wir aufklären, wie Mykobakterien den Lipidstoffwechsel und die Lipidtransportwege des Wirts manipulieren, möchten wir neue therapeutische Angriffspunkte für TB und andere intrazelluläre Infektionen identifizieren. Durch das Blockieren von Lipidversorgungspfaden oder das Stören der Interaktion von Lipidtröpfchen mit dem Erreger könnten Wirtsabwehrmechanismen gestärkt und bakterielle Überlebensstrategien geschwächt werden.
Aktuelle Projekthighlights
- Charakterisierung von Fettsäure-aktivierenden Enzymen in Mykobakterien und Analyse ihrer Rolle bei Lipidversorgung, Wirts-Pathogen-Lipidflüssen und dem Vakuolenescape.
- Untersuchung von Membrankontaktstellen (MCSs) zwischen dem endoplasmatischen Retikulum (ER) und pathogenhaltigen Vakuolen, um zu verstehen, wie diese den Lipidtransport zu Mykobakterien ermöglichen.
- Entwicklung genetisch modifizierter humaner Makrophagenmodelle, um Dynamiken von Lipidtröpfchen, „nutritional immunity“ und das Überleben von Pathogenen in humanen Wirtszellen zu beobachten.
Ausblick
Aufbauend auf den mechanistischen Erkenntnissen aus Dictyostelium discoideum und Mycobacterium marinum möchten wir nun unsere Werkzeuge, Bildgebungs-Pipelines, Lipid-Tracking-Sonden und analytischen Workflows auf Mycobacterium tuberculosis-Infektionen übertragen.
Dieser strategische Schritt ermöglicht es uns, zentrale Wirts-Pathogen-Interaktionswege direkt im humanen TB-Erreger zu validieren und therapeutisch relevante lipidabhängige Schwachstellen zu identifizieren.
Karriere & Kontakt
Wir freuen uns über Bewerbungen von motivierten Master- und Promotionsstudierenden sowie Postdocs, die Interesse an Infektionsbiologie, Wirts-Pathogen-Interaktion, Lipidmetabolismus und Mikroskopie haben.
Bitte kontaktieren Sie uns per E-Mail mit einem kurzen Lebenslauf und einer Darstellung Ihrer Forschungsinteressen.
Projekt A: Fettsäure-Aktivierung und mykobakterielles Überleben
Dieses Projekt untersucht die Rolle fettsäureaktivierender Enzyme in pathogenen Mykobakterien und wie Veränderungen im Fettsäurestoffwechsel von Wirt und Erreger die Lipidversorgung, den Vakuolenescape und das intrazelluläre Überleben beeinflussen. Durch den Einsatz biochemischer Lipidmethoden, fortgeschrittener Lebenzellmikroskopie, Lipidomics und unterschiedlicher Infektionsmodelle wollen wir aufklären, wie Mykobakterien die Lipidnetzwerke des Wirts ausnutzen und neue Enzyme für therapeutische Interventionen identifizieren.
Projekt B: Lipidtröpfchen-Interaktionen während der mykobakteriellen Infektion
Lipidtröpfchen (LDs) in Wirtszellen werden von intrazellulären Mykobakterien umfunktioniert, um die Produktion von Speicherslipiden und das bakterielle Überleben zu unterstützen. Mithilfe von Lebendzellmikroskopie, cryo-CLEM (korrelative Licht-/Elektronenmikroskopie), tomographischer Bildgebung und Makrophagenmodellen untersuchen wir, wie LDs rekrutiert, verändert und ihre Lipide an Pathogenkompartimente übertragen werden. Ziel ist es, die räumlich-zeitliche Dynamik der LD-Pathogen-Interaktion zu kartieren und Wirtsstrategien der Lipidsequestrierung als „nutritional immunity“ zu bewerten.
Langfristig möchten wir die identifizierten LD-zu-Pathogen-Lipidtransferwege als potenzielle Transportmechanismen nutzen, um hydrophobe antimikrobielle Wirkstoffe direkt an intrazelluläre Mykobakterien zu liefern.
Projekt C: Membrankontaktstellen und Lipidtransfer während der Infektion
Intrazelluläre Mykobakterien manipulieren Wirtsmembranen, um Zugang zu Lipiden zu erhalten. Unsere Arbeit konzentriert sich auf ER-abgeleitete Membrankontaktstellen (MCSs), die sich zwischen dem endoplasmatischen Retikulum und der mykobakterienhaltigen Vakuole (MCV) bilden. Mittels fortgeschrittener Bildgebungstechniken wie Serial-Block-Face-SEM sowie funktioneller Assays wollen wir die molekulare Architektur und funktionelle Relevanz dieser MCSs im Lipidtransfer, bei der Reparatur beschädigter Vakuolen und beim Escape des Erregers definieren.
Projekt D: Translationale humanzelluläre Infektionsmodelle & therapeutische Ansätze
Um mechanistische Erkenntnisse mit biomedizinischer Relevanz zu verknüpfen, entwickeln wir genetisch modifizierte humane Makrophagensysteme, die Infektionsdynamiken, Lipidsignale, Organelleninteraktionen und das Überleben des Erregers erfassen. Parallel dazu erforschen wir neue therapeutische Strategien, die die einzigartige lipidreiche Zellhülle von Mykobakterien angreifen, darunter antimikrobielle Peptide und host-directed Inhibitoren von Lipidwegen.
Darüber hinaus entwickeln wir diagnostische Ansätze, die Lipidsignaturen oder lipidassoziierte Wirtsantworten nutzen, um mykobakterielle Infektionen früher und präziser nachweisen zu können. Dieses Projekt baut auf Erkenntnissen aus Modellorganismen auf und zielt auf einen translationalen Beitrag zur TB- und intrazellulären Infektionsforschung.
Ausblick: Werkzeugtransfer auf Mycobacterium tuberculosis
Aufbauend auf Erkenntnissen aus Modellsystemen werden wir in der nächsten Phase unsere Werkzeuge – Bildgebungspipelines, funktionelle Lipidsonden, Lipidfluss-Analysen und Infektions-Workflows – direkt auf Mycobacterium tuberculosis übertragen. Dies ermöglicht eine präzise Validierung zentraler Wirts-Pathogen-Lipidinteraktionswege im humanen TB-Erreger und eröffnet neue Möglichkeiten zur Identifizierung lipidabhängiger therapeutischer Angriffspunkte.
- 2019 – 2022
SFB944 – P25: „Funktionelle Auswirkungen der Lipidlogistik auf die Infektion mit Mykobakterien in Dictyostelium“ @Uni Osnabrück - 2020 – 2023
SPP2225: „Funktionelle Auswirkungen von mykobakteriellen Lipiden auf die Infektion von Dictyostelium mit M. marinum“ - 2023 – 2026
SFP1557 – P1: „Manipulation und Ausnutzung des Lipidtransfers durch pathogene Mykobakterien“ @Uni Osnabrück
- Lebendzellmikrokopie (Spinning Disc und Lattice Lightsheet)
- Klassische Immunofluoreszenz
- Elektronenmikroskopie
- Kryo-Tomographie
- Korrelative Licht- und Elektronenmikroskopie
- Expansionsmikroskopie
- Dünnschichtchromatographie
- Massenspektrometrie (Lipidomik und Proteomik)

2024
Listian, SA, Mazur, A-C, Kol, M, Ufelmann, E, Eising, S, Fröhlich, F, Walter, S, Holthuis, JCM & Barisch, C 2024, 'Complex sphingolipid profiling and identification of an inositol-phosphorylceramide synthase in Dictyostelium discoideum', iScience, Jg. 27, Nr. 9, S. 110609. https://doi.org/10.1016/j.isci.2024.110609
2023
Anand, A, Mazur, A-C, Rosell-Arevalo, P, Franzkoch, R, Breitsprecher, L, Listian, SA, Hüttel, SV, Müller, D, Schäfer, DG, Vormittag, S, Hilbi, H, Maniak, M, Gutierrez, MG & Barisch, C 2023, 'ER-dependent membrane repair of mycobacteria-induced vacuole damage', mBio, Jg. 14, Nr. 5, S. e0094323. https://doi.org/10.1128/mbio.00943-23
Barisch, C, Holthuis, JCM & Cosentino, K 2023, 'Membrane damage and repair: a thin line between life and death.', BIOLOGICAL CHEMISTRY . https://doi.org/0.1515/hsz-2022-0321
Franzkoch, R*, Anand, A*, Breitsprecher, L, Psathaki, OE & Barisch, C 2024, 'Resolving exit strategies of mycobacteria in Dictyostelium discoideum by combining high-pressure freezing with 3D-correlative light and electron microscopy', MOLECULAR MICROBIOLOGY, Jg. 121, Nr. 3, S. 593-604. https://doi.org/10.1111/mmi.15205 *authors contributed equally
Vormittag, S, Hüsler, D, Haneburger, I, Kroniger, T, Anand, A, Prantl, M, Barisch, C, Maaß, S, Becher, D, Letourneur, F & Hilbi, H 2023, 'Legionella- and host-driven lipid flux at LCV-ER membrane contact sites promotes vacuole remodeling', EMBO REPORTS , Jg. 24, Nr. 3, e56007. https://doi.org/10.15252/embr.202256007
Additional publications of Caroline Barisch not affiliated with the Research Center Borstel
2026
Foulon, M, Huettel, SV, Breitsprecher, L, Gauda, W, Raykov, L, Koliwer-Brandl, H, Ohlhagen, M, Thuenauer, R, Schwudke, D, Hilbi, H, Soldati, T & Barisch, C, Intracellular compartmentalization shapes lipid access and metabolic fitness of mycobacteria, 2026, bioRxiv. https://doi.org/10.64898/2026.02.10.704997

Leitung der Forschungsgruppe

Mitarbeiter:innen











Zentrum für strukturelle Systembiolgie (CSSB)
DESY, Gebäude 15
Notkestraße 85
D-22607 Hamburg
Assoziiert mit dem Fachbereich Biologie & der MIN-Fakultät der Universität Hamburg
Tel: +49 (0)40-8998-87620
https://www.barischlabcssb.com
Alumni
- Erasmus student MSc Laura Antón Esteban
- BSc Michelle Hacker
- MSc Iris Hube
- BSc Fynn Kamp
- Dr. Stevanus Listian
- MSc Danica Müller
- Josephina Offt
- BSc Sarah Polkehn
- MSc Deise Schäfer
- MSc Edwin Ufelmann
- MSc Hritik Yadav