Klinische und Molekulare Allergologie

(Anmeldung für Patienten: Tel.: 0451-500-44165; -44195; -44196 sowie unter der E-Mailadresse ambulanz.innere.luebeck@uksh.de)
Die Forschungsgruppe Klinische und Molekulare Allergologie arbeitet an der Aufklärung von Pathomechanismen der Allergie- und Asthmaentstehung und möglicher Sensibilisierungswege. Dazu werden neue Allergene sowie Allergie-relevante Epitope aus verschiedenen Allergenquellen (Inhalationsallergenquellen, Nahrungsmittel und Biologika) identifiziert und hinsichtlich ihrer molekularen Struktur und deren Einfluss auf ihre Funktion und Allergenität charakterisiert. Durch die enge Anbindung der Forschungsgruppe an die Interdisziplinäre Allergie-Ambulanz, Universität zu Lübeck kommen neueste Forschungsergebnisse den Patienten zugute (Translation), und es kann die Grundlagenforschung mit Primärmaterial klinisch gut charakterisierter Patienten in Ethik-gestützten Studien durchgeführt werden. Auf diese Weise sollen Markerallergene identifiziert und Testverfahren entwickelt werden, die die Allergiediagnostik zielgenauer gestalten. Dadurch läßt sich nicht nur das individuelle Risiko einzelner Betroffener, schwer zu reagieren, erfassen, sondern in Zukunft auch eine effektivere und sicherere Therapieentscheidung treffen. Die neuen Erkenntnisse zu Struktur und Funktion stellen zudem eine Basis für die Erarbeitung neuer Therapieoptionen dar.
Hinweis
Es besteht die Möglichkeit zur Anfertigung von Masterarbeiten für Studentinnen/Studenten der Studiengänge Biologie, Biochemie, Lebensmittelchemie, Ernährungswissenschaften sowie verwandter Naturwissenschaften. Informationen finden Sie unter der Rubrik Projekte. In diesen Bereichen werden ebenfalls Dissertationen vergeben – Bitte Ausschreibungen beachten.
Allgemeines
Allergien sind komplexe Multiorganerkrankungen, deren Häufigkeit rapide zunimmt. 20-30 Mio. Menschen in Deutschland haben eine allergische Erkrankung (ohne Asthma). Damit besitzen Allergien den Status einer „Volkskrankheit“. Einige Allergien sind lebensbedrohlich oder Wegbereiter für chronische Atemwegserkrankungen wie z.B. die Entwicklung des Asthma bronchiale aus einer vorbestehenden allergischen Rhinitis (z.B. „Heuschnupfen“) im Sinne des „Etagenwechsels“. Sie sind chronisch-rezidivierend (wie die allergische Rhinokonjunktivitis) und führen somit zur Minderung der Lebensqualität sowie zu erheblichen Leistungseinschränkungen und dadurch nicht zuletzt zu großen sozio-ökonomischen Belastungen (Weißbuch Allergologie in Deutschland 2025).
Das Krankheitsbild der Allergie entsteht durch eine überschießende Reaktion des Immunsystems gegen an sich harmlose Substanzen natürlichen Ursprungs. Allergien werden sowohl von genetischen Einflüssen als auch von Umwelteinflüssen geprägt.
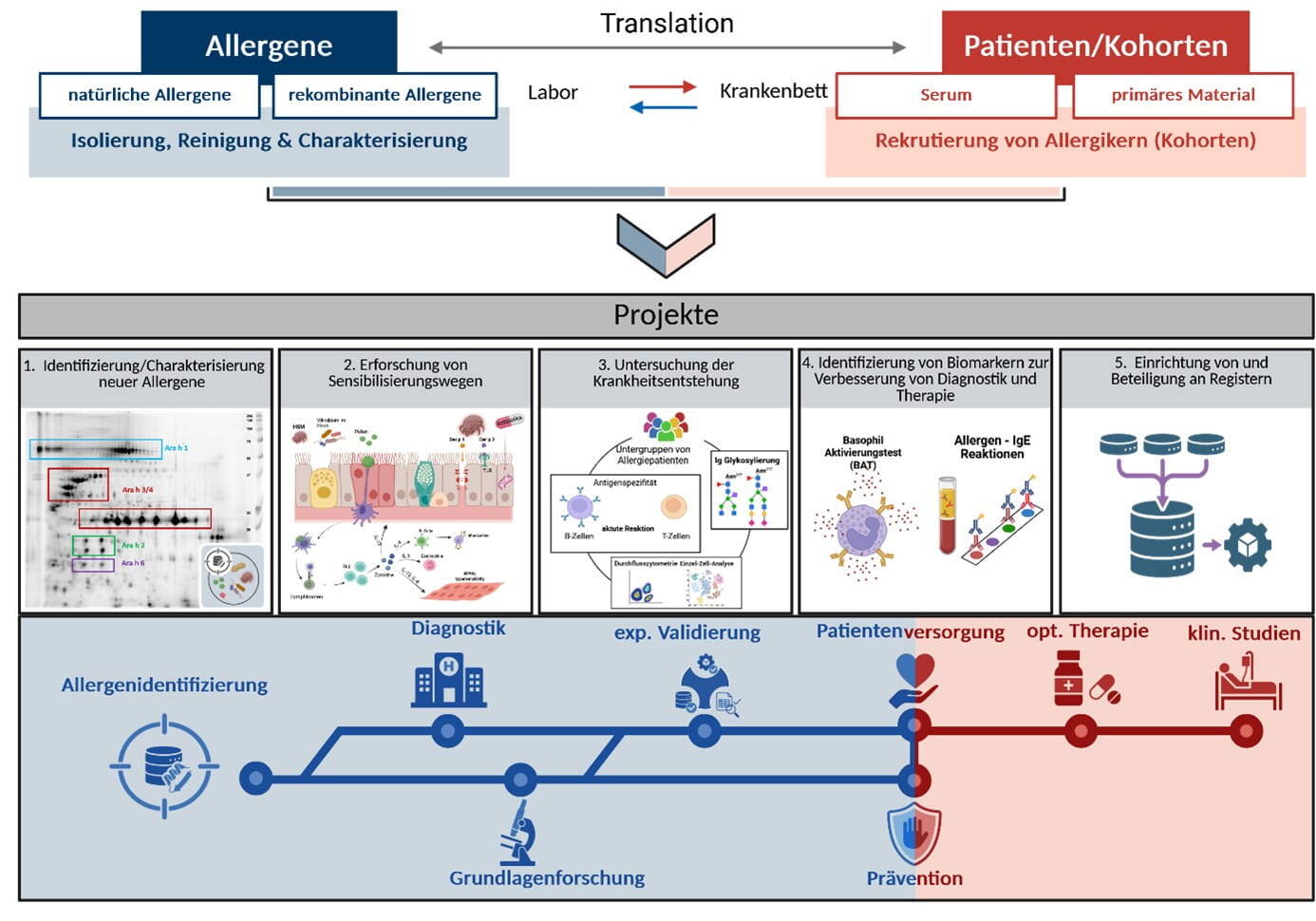
Die Forschungsgruppe (FG) Klinische und Molekulare Allergologie hat die folgenden Schwerpunkte (siehe Abb. 1):
- Den Einfluss der Allergene auf die Sensibilisierung(swege).
- Die Entwicklung von Allergien und Asthma (z.B. mittels Trained Immunity).
- Die schrittweise Änderung der Organbezogenheit („Etagenwechsel“),
- Die Schwere sowie Lokalisation der allergischen Symptomausprägung (Lunge, Gastrointestinaltrakt, Haut, etc.), mit der sogenannten „Molekularen Phänotypisierung“.
Anhand von Markerallergenen und individueller Sensibilisierungsprofile in unseren und anderen Patientenkohorten möchten wir folgende Forschungsziele erreichen:
- Das Verständnis der Allergie- und Asthmaentstehung zu verbessern.
- Das Wissen über Krankheitsentwicklung/-verlauf (Trajektorien allergischer Erkrankungen) zu erweitern.
- Eine Vorhersage des Schweregrads der Allergie zu ermöglichen.
- Eine Unterscheidung von Asthmaphänotypen zu vereinfachen.
- Die Verbesserung der Therapieentscheidung (Erfassung des „Theratyps“)
- Eine Vorhersage des Therapieerfolgs treffen zu können.
Projekte der Forschungsgruppe (FG) Klinische und Molekulare Allergologie
1. Identifizierung/Charakterisierung neuer Allergene (CARE)
Die Identifizierung neuer Allergene ist ein Kernpunkt unserer Arbeiten und dient dazu, Biomarker für die Schwere der Erkrankung sowie die Organbezogenheit für die klinische Diagnostik zu etablieren. Die Charakterisierung der Proteine hinsichtlich Struktur, Molekulargewicht, hydrophiler/lipophiler Eigenschaften, Funktion (z. B. Protease) und Patienten-IgE Bindung soll dabei helfen, die Frage zu klären: Was macht ein Molekül zum Allergen? Was sind relevante unterstützende Faktoren?
So gelang es unserer FG bereits, Einzelallergene aus verschiedenen Nahrungsmitteln pflanzlichen Ursprungs zu identifizieren und zu isolieren, die als Markerallergene mit dem Schweregrad der Symptomatik assoziiert zu sein scheinen. Zu den identifizierten Allergenen zählen: das Lipidtransferprotein (LTP) der Haselnuss Cor a 8, aus der gelben Lupine sowie aus der blauen Lupine (Lup an 3) das Profilin aus der weißen Lupine (Lup a 5), das Speicherprotein der Erdnuss Ara h 6, das LTP der Erdnuss Ara h 9, die Erdnuss-Defensine Ara h 12 und Ara h 13, sowie die Oleosine der Erdnuss Ara h 14 und Ara h 15. Untersuchungen mit dem Blut von Patienten mit leichter und schwergradiger Erdnussallergie zeigten bereits, dass nur die Patienten mit schwerer Erdnussallergie IgE-Antikörper gegen die Oleosine aufweisen.
Die isolierten und charakterisierten Allergene sind wichtige Ausgangspunkte für die
- Aufklärung von Sensibilisierungswegen (CONTROL)
- Aufklärung der Krankheitsentstehung und der Krankheitsdynamik (CONTROL)
- Identifizierung von Biomarkern zur Verbesserung der Diagnostik und Therapie (CARE)
2. Aufklärung von Sensibilisierungswegen (CONTROL)
Die Entstehung der Sensibilisierung gegen bestimmte Proteine und die sich daraus entwickelnden Symptome bzw. Allergie/n sind noch nicht aufgeklärt. Die Erdnussallergie ist aufgrund der Schwere der allergischen Reaktion bei geringem Allergenkontakt eine hierfür sehr geeignete „Modellerkrankung“.
Die Sensibilisierung bei Nahrungsmittelallergien kann über verschiedene Grenzflächen erfolgen: a) Den Verdauungstrakt (Stillen und oral über die Ernährung), b) die Haut (für die Erdnussallergie derzeit der favorisierte Sensibilisierungsweg), c) den Atmungstrakt oder bereits d) im Mutterleib (in utero).
So konnten wir zeigen, dass die Markerallergene der Erdnuss Ara h 2 und 6 (Speicherproteine) über die Nahrung in die Muttermilch gelangen.
In der Allergologie ist nun die Frage bedeutsam, ob eine Erdnussexposition des Säuglings in der Stillzeit einen Sensibilisierungsweg oder möglicherweise sogar einen Weg zur Toleranzinduktion darstellt.
Wir konnten bereits mehrere neue Erdnusseinzelallergene in unserer FG identifizieren und charakterisieren. Da einige Erdnussallergene umweltstabil sind und über die Luft vermittelt werden können (Auslösen z.B. eines Bronchospasmus bei schweren Erdnussallergikern nach Öffnen einer Erdnussflipstüte aus einiger Entfernung), verwenden wir diese als Werkzeuge in Projekten des Deutschen Zentrums für Lungenforschung (DZL) zur mechanistischen Aufklärung der oben genannten Prozesse am Atemtrakt.
Ein besonderer Fokus unserer Arbeit liegt dabei auf der Interaktion von lipophilen Allergenen und den Lipiden der entsprechenden Allergenquelle (Matrix) sowie ihrem Einfluss auf die Allergenität. Beispiele solcher Allergene sind Oleosine, Lipidtransferproteine aus pflanzlichen Quellen und die Galaktose-alpha 1,3-Galaktose (Alpha-Gal) aus Säugetierfleisch. Des Weiteren wird untersucht, ob und mittels welcher verschiedenen Mechanismen lipophile Allergene für schwere allergische Reaktionen bzw. die Asthmaentstehung verantwortlich sind (Flagship Project Basic Science des DZL; Koordination: Prof. Dr. med. Uta Jappe).
Neben Einzelallergenen aus der Erdnuss (die Erdnussallergie ist eine der am schwersten verlaufenden Nahrungsmittelallergien) werden Einzelallergene der Inhalationsallergenquelle Hausstaubmilbe zur Erfassung individueller Sensibilisierungsprofile rekombinant hergestellt und in neu entwickelten, serumsparenden IgE-Nachweismethoden eingesetzt. Diese Tests werden es ermöglichen, bereits bei ganz kleinen Kindern aus Allergikerfamilien, von denen naturgemäß wenig Blut gewonnen werden kann (z.B. DZL-ALLIANCE-Kohorte), diejenigen Einzelallergene zu identifizieren, die als „Initiatorallergene“ die allerersten Sensibilisierungen auslösen. Das ist nicht nur für das Verständnis der Erkrankung und Krankheitstrajektorien, sondern auch für die Entwicklung von Therapiekonzepten bzw. die Entscheidung, welche Patienten von einer Therapie profitieren und welche voraussichtlich nicht, von größter Bedeutung (Abb. 1, Punkt 2 und Punkt 4).
3. Aufklärung der Krankheitsentstehung (CONTROL)
Entscheidend für die Aufklärung des Pathomechanismus der Soforttyp-(Typ I)-Allergie ist es herauszufinden, wie es zur individuellen Auslösung der Bildung bestimmter Allergie-vermittelnder Antikörper vom IgE-Typ durch die einzelnen Allergene kommt. Dazu wird unter anderem die spezifische Interaktion von Allergenen an Grenzflächen (Oberflächenepithelien des Atmungs- bzw. Gastrointestinaltrakts) untersucht. Ziel ist es aufzuklären, wie die Verstoffwechselung der Allergene erfolgt, ob es hierbei Unterschiede zwischen Allergikern und Nichtallergikern gibt und wie es zur Fehlregulierung und damit zur Sensibilisierung und schließlich zur allergischen Reaktion kommt. Die Aufklärung der Pathomechanismen ist die rationale Vorgehensweise, um die Krankheit Allergie zu bekämpfen, aber auch Vorbeugung betreiben zu können (Abb. 1, Punkt 3).
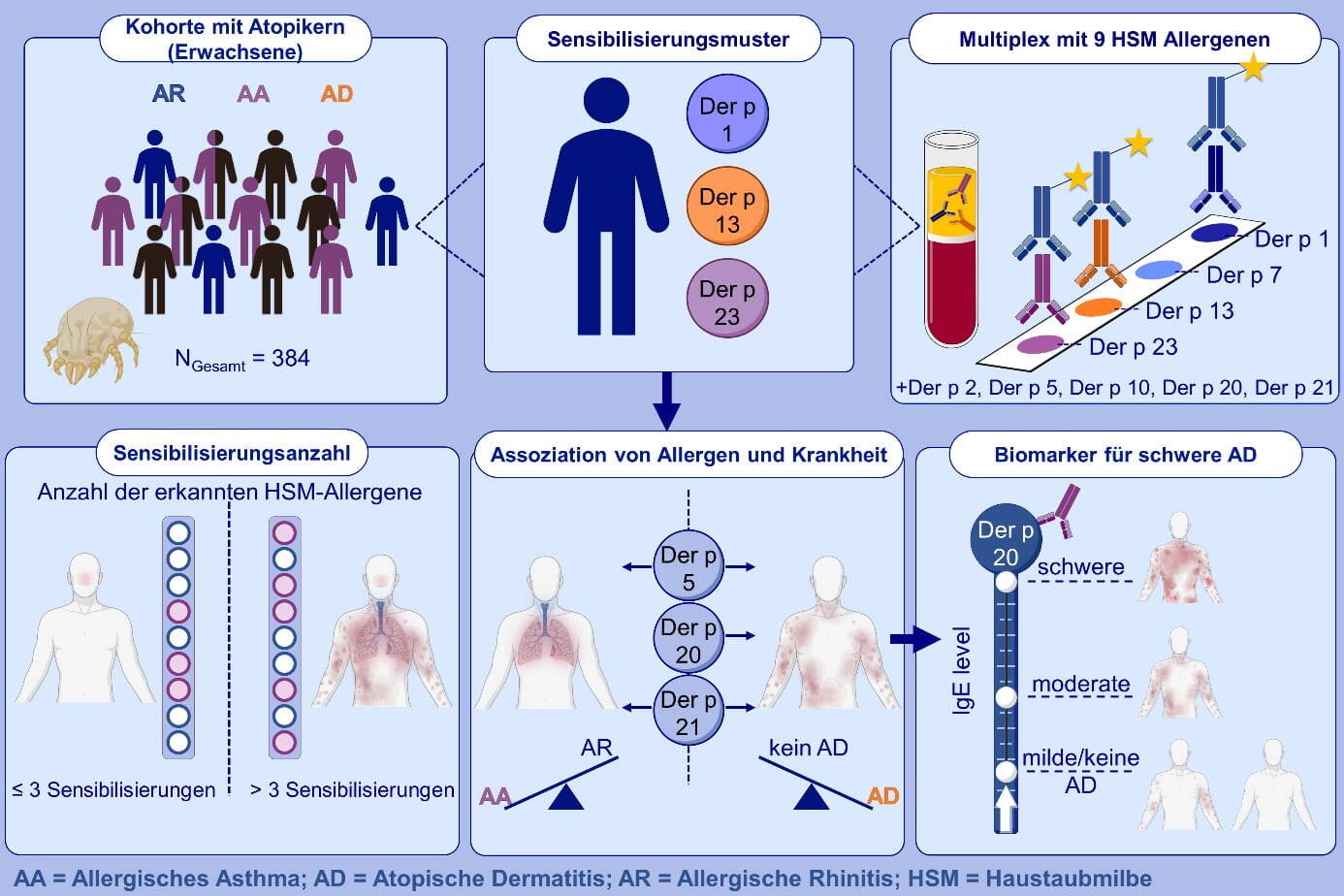
So konnte in der Gruppe gezeigt werden, dass Hausstaubmilbenallergiker mit unterschiedlichen Phänotypen individuell unterschiedliche Sensibilisierungsprofile aufwiesen (Abb. 2.). Entsprechende Allergene wurden rekombinant in E. coli hergestellt und gereinigt. Mit Hilfe eines Multiplex-Assays mit verschiedenen Allergenen konnten Seren von Patienten auf ihre IgE-Reaktivität untersucht werden. Die Seren von verschiedenen Probandengruppen (Rhinitis, allergisches Asthma und atopische Dermatitis) zeigten hierbei unterschiedliche Reaktionsmuster gegenüber den Allergenen, wobei Patienten mit Asthma auf mehr Allergene reagierten als Patienten mit Rhinitis. Deutliche Unterschiede zeigen sich hierbei für Der p 5 und Der p 21, sodass diese als Markerallergene für das Hausstaubmilben-assoziierten Asthma genannt werden können.
Im Vergleich konnten wir bei der atopischen Dermatitis deutlich mehr Sensibilisierungen gegen Hausstaubmilbenallergene beobachten. Darunter waren IgE-Reaktionen auf Der p 5, Der p 21, aber auch auf Der p 10, Der p 13 und Der p 20. Aufgrund dieser Ergebnisse wird aktuell die Organspezifität der Hausstaubmilbenallergene in einer Kooperation mit der Medizinischen Hochschule Hannover (MHH) im Rahmen eines DFG-Projektes (JA 1007/4-1) untersucht.
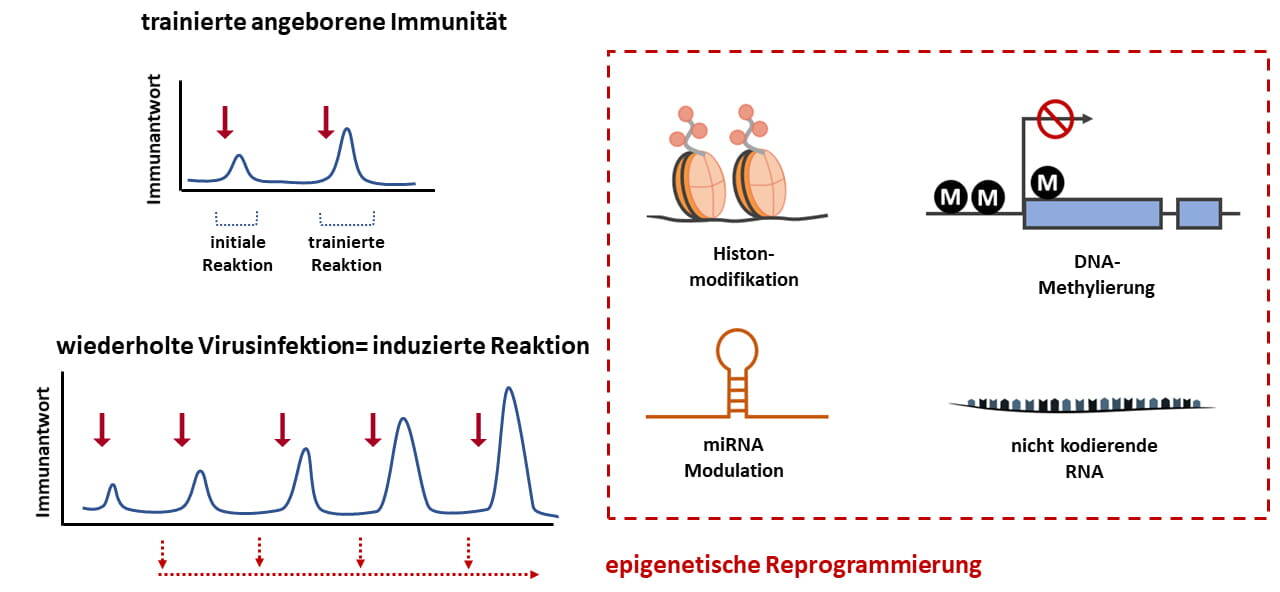
Trained Immunity (Control)
Seit den letzten drei Jahren arbeiten wir zudem – unterstützt durch DZL-Fördermittel – an der Aufklärung der Mechanismen der trainierten Immunität in Epithelien und ihres Einflusses auf die Allergieentstehung. Wir haben untersucht, wie Epithelzellen, die lange Zeit als passive physikalische Barrieren galten, nach einer Infektion gedächtnisähnliche Eigenschaften erwerben können. Ergebnisse dieses Projekts zeigen, dass wiederholte Virusinfektionen eine dauerhafte Umprogrammierung des Epithels bewirken und damit die Reaktion der Epithelzellen auf nachfolgende Herausforderungen verändern können (Abb. 3). Auf dieser Grundlage ist es unser nächstes Ziel, den mechanistischen Zusammenhang zwischen mikrobieller Exposition und der Umprogrammierung von Epithelzellen im Zusammenhang mit chronischen Entzündungskrankheiten zu definieren. Diese Forschungsrichtung wird klären, wie mikrobielle Expositionen in der frühen Kindheit oder beim ersten Kontakt das Verhalten von Epithelzellen bei späteren „Second-Hit“-Ereignissen, wie z. B. Allergenexposition bzw. weitere Auslöser von Asthma, beeinflussen. Mit dieser Arbeit wollen wir ein tieferes Verständnis dafür gewinnen, wie das epitheliale Gedächtnis zur Anfälligkeit für Krankheiten und deren Fortschreiten beiträgt („epitheliale Immunität“).
4. Identifizierung von Biomarkern zur Verbesserung der Diagnostik und Therapie (CARE)
Die Identifikation von Biomarkern im Serum von allergischen Patienten ist ein weiteres wichtiges Forschungsfeld unserer Gruppe. Diese Biomarker sind beispielsweise a) verstoffwechselte und/oder immunologisch veränderte Allergene, b) Allergen-gekoppelte Trägermoleküle, c) Antikörper vom Typ IgE, aber auch d) andere Antikörperklassen (IgG, IgA, etc.), die gegen Allergene gerichtet sind. Dabei können bestimmte IgE-bindende Motive als Risikofaktoren für die Schwere der Symptomausprägung dienen oder auch ein bestimmtes (IgE-assoziiertes) Antikörper-Risikoprofil des Patienten. Als Beispiel gilt für Erdnussallergiker z. B. IgE-Positivität für die Speicherproteine Ara h 1, Ara h 2 und Ara h 3 sowie für die Oleosine Ara h 10, Ara h 11, Ara h 14 und Ara h 15 (Abb. 1, Punkt 4). Der Nachweis von IgE-Antikörpern gegen Einzelallergene stellt eine molekülspezifische Diagnostik dar, deren Aussagefähigkeit - je nach Allergenquelle und klinischer Charakterisierung der Einzelallergene - zu klären ist. Dieses wiederum ist nur möglich durch die enge klinische Anbindung der Forschungsgruppe an die von Frau Prof. Dr. med. Uta Jappe zusätzlich in Personalunion geleiteten Interdisziplinären Allergie-Ambulanz am UKSH Campus Lübeck an der Universität zu Lübeck (UzL).
Ein durchflusszytometrischer Basophilen-Aktivierungstest (BAT), der zwischen allergisch und sensibilisiert differenziert, wurde in Kooperation mit der Zentralen Einheit Fluoreszenz-Zytometrie des FZB optimiert und sowohl die Gating-Strategie (EP4200610A1 und US20230349890A1) als auch ein BAT-basierter Algorithmus (2023) als Patente (WO2025/003040) angemeldet. Aktuell läuft hierzu eine klinische Untersuchung bei allergischen Kindern in Kooperation mit der Klinik für Dermatologie der Elbe Kliniken Buxtehude (Studie 10-126).
Optimierung der Individualisierten Diagnostik
Die Identifizierung und Charakterisierung relevanter Einzelallergene, die Klonierung und Produktion als rekombinante Allergene sowie die Synthese Epitop-darstellender Peptide ermöglicht eine Komponenten-aufgelöste Diagnostik. Dadurch können IgE-Antikörper, die für ein allergenes Protein oder ein lineares Epitop spezifisch sind, nachgewiesen werden. So lassen sich individuelle Sensibilisierungsmuster (Allergogramme) der Patienten gegen verschiedene a) Proteine z. B. allergener Nahrungsmittel (Erdnuss, Lupine, Weizen, Fleisch, etc.), Hausstaubmilbenallergene, Pollen, Medikamente (Biologika), etc., gegen b) homologe, d.h. strukturverwandte, Proteine in verschiedenen Nahrungsmitteln oder gegen c) verschiedene Antikörper-bindende Motive (IgE-Bindungsstellen) eines einzelnen Allergenmoleküls erstellen (Präzisionsdiagnostik). Mit den Oleosinen aus der Erdnuss wurden Markerallergene für die Schwere der allergischen Reaktion isoliert und charakterisiert. Da diese Allergene nicht wasserlöslich sind, kommen sie in den bisherigen Diagnostik- und (höchstwahrscheinlich) Therapie-Lösungen nicht vor. Wir arbeiten derzeit daran, sie für die Routinediagnostik zugänglich zu machen (Transfer) und überprüfen parallel ihren Einsatz in Therapiemodellen (Abb. 4).
Weiterhin können wir den Patienten in unseren Allergie-Ambulanzen inzwischen eine empfindliche Multianalyten-Diagnostik zur Abklärung der verzögerten Anaphylaxie auf Säugetier-Fleisch (Alpha-GAL-Syndrom) anbieten. In einigen Fällen von Alpha-GAL-Syndrom zeigte diese Diagnostik einen entscheidenden Vorteil gegenüber dem Routineverfahren, um die Allergie nachzuweisen (Präzisionsdiagnostik). Dadurch konnten die Patienten ihre Ernährung anpassen und somit ihr Risiko auf Anaphylaxie reduzieren.
In einem Proof-of-Principle Projekt konnten wir Epitope auf Biologika (Therapieantikörper) entdecken, die verantwortlich für den Wirkverlust zu sein scheinen. Damit wurde die Voraussetzung dafür geschaffen, rechtzeitig das Biologikum zu wechseln und eine Alternative zu wählen, die sicher UND wirksam ist, bei der es also nicht durch Kreuzreaktivität über Allergengemeinsamkeiten von vornherein zu Wirkverlust oder Hyperreaktivität kommt (individualisierte Diagnostik, Präzisionsdiagnostik) (Abb. 1, Punkt 4).
ImproviNg DIagnostiCs And ThErapy of Food Hypersensitivity (INDICATE-FH): BMBF-Verbundprojekt
Seit 2021 arbeiten wir in Kollaboration mit dem Institut für Ernährungsmedizin des Universitätsklinikums Schleswig-Holstein (Universität Lübeck) und der Universität Hohenheim an der Verbesserung der Diagnostik der allergisch und nicht-allergisch bedingten Weizenunverträglichkeit: BMBF-Projekt "Neue Wege in der Diagnostik und Therapie von Nahrungsmittelunverträglichkeiten", im Englischen "ImproviNg DIagnostiCs And ThErapy of Food Hypersensitivity" (INDICATE-FH). Nahrungsmittelunverträglichkeiten gegenüber Weizenmehl sind in der medizinischen Praxis häufig auftretende Probleme. In vielen Fällen werden die unspezifischen gastrointestinalen Symptome als Reizdarmsyndrom fehlinterpretiert. Hauptursache hierfür ist das Fehlen von spezifischen Biomarkern und praxistauglichen Diagnoseverfahren. Das Hauptziel des Projekts ist es, die bestehende Diagnostik für Nicht-Zöliakie-Weizensensitivität (NCWS) in Abgrenzung zu Zöliakie (CD), Weizenallergie (WA) und zum Reizdarmsyndrom zu optimieren. Die FG Klinische und Molekulare Allergologie befasst sich mit der Pathogenese von und Verbesserung der Diagnostik für Weizenallergie, indem zunächst allergene Proteine aus Weizen und Lupine identifiziert, gereinigt, charakterisiert und rekombinant hergestellt werden, um die Sensitivität und Spezifität von Allergietests zu verbessern. Diese werden schließlich in Antikörper- und Zell-basierte Assays eingesetzt, mit dem Ziel, zukünftig sicherer die WA von CD und NCWS sowie Darmerkrankungen anderer Genese unterscheiden zu können. Zusätzlich werden die Einzelallergene daraufhin untersucht, ob sie im Sinne eines Biomarkers mit der Schwere einer allergischen Reaktion assoziiert sind. Darüber hinaus sollen mittels Transkriptom- und Methylom-Analysen des Bluts Gene mit Biomarker-Potenzial identifiziert werden, die in Zukunft die Unterscheidung zwischen allergischen und nicht-allergischen Reaktionen auf Weizen anhand eines einfachen Bluttests ermöglichen könnten (Kooperation mit Prof. T. Goldmann, Borstel). Die rekombinanten Einzelallergene des Weizens dienen darüber hinaus zur mechanistischen Pathogeneseaufklärung mittels Zellkultur. Dadurch wird die Diagnose und - letztendlich - die Behandlung von Patienten mit Symptomen auf Weizenprodukte optimiert, was für viele Patienten eine Erleichterung darstellt, da sie ihre Ernährung entsprechend umstellen können, um möglichst beschwerdefrei durch den Alltag zu kommen.
Zukunftsperspektiven
In den nächsten Jahren werden unsere lipophilen Allergene in Routinediagnostikplattformen überführt.
Wir werden genaueres über die Organspezifität bestimmter einzelner Allergene wissen und die Mechanismen, über die sie direkt am Epithel aber auch in den Immunzellen wirken.
Für die Diagnostik der Weizenallergie werden uns zukünftig weitere Allergene zur Verfügung stehen, aber auch die Diagnose anderer Weizen-assoziierter Erkrankungen soll mittels nicht-Allergen-, aber –omics-basierter Tests zukünftig besser möglich werden.
Die Aufklärung der Wechselwirkung mikrobieller Komponenten mit einzelnen Allergenen und ihre Rolle bei der Allergieentstehung im Rahmen der trainierten Immunität wird uns neue Wege zur Primärprävention aufzeigen.
In diesem Zusammenhang werden wir zukünftig stärker auf mikrobielle Allergene fokussieren, die direkt Asthma und Allergien auslösen, aber auch eine Brücke zwischen postinfektiöser und allergischer Entzündung darstellen können.
5. Aufbau von Registern und Datenbanken
Die Daten, die im Rahmen von Registern und Datenbanken gesammelt werden (Abb. 1, Punkt 5), finden Eingang in epidemiologische Studien und dienen der Überwachung im Hinblick auf das Auftreten von neuen Allergien (Deutsches und Europäisches Anaphylaxie-Register, NORA). Des Weiteren arbeitet die Forschungsgruppe mit der BioMaterialBank (BMB) Nord zusammen.
Deutsche Forschungsgemeinschaft
- DFG-Antrag JA 1007/4-1 - Die Rolle von Hausstaubmilben-Einzelallergenen - strukturelle und funktionelle Besonderheiten im Kontext atopischer Erkrankungen 2023-2027
- DFG JA 1007/2-3: Investigation of peanut oil bodies and the new lipophilic allergens like oleosins regarding promotion of sensitization and elicitation processes in food allergy (2019-2022)
- DFG JA1007/2-1: Struktur-und Funktionsuntersuchungen von lipophilen Erdnussallergenen insbesondere den Olesosinen (2013-2015)
- DFG Scho 828/2-1: Untersuchung von Erdnussantigenen/-allergenen in der Muttermilch nach Prozesierung an den Grenzflächen und ihre Interaktion mit dem Immunsystem
Exzellenz Cluster: 2019 - 2032 Precision Medicine in Chronic Infammation (PMI)
Bundesministerium für Bildung und Forschung
- FKZ:01EA2109B Verbundprojekt „Neue Wege in der Diagnostik und Therapie von Nahrungsmittelunverträglichkeiten; INDICATE-FH“ (PI: U. Jappe) (2021-2024)
- DZL: Deutsches Zentrum für Lungenforschung (DZL), Research Center North
(ARCN) (PI: U. Jappe), beteiligt an: Disease Area Asthma/Allergy; AA-FPB: Koordination des Flagship Projekts Basic Science: U. Jappe (2010-2027) - DZL - Flexible Funds; Single Cell Analysis (SCA): Cell Circuits in Predictive and Personalized Pulmonary Medicine. PI Prof. Uta Jappe, cooperation with Prof. Marc Ehlers, UzL (2024-2027)
- FK 01KG 0911 Verbundprojekt „Birch Associated Soy Allergy and Immuno-Therapy; BASALIT (PI. U. Jappe) (2009-2015)
Bundesministerium für Wirtschaft und Technologie
- ZIM-KF2784702SB4: Förderprogramm Zentrales Innovationsprogramm Mittelstand (ZIM): „Entwicklung eines innovativen Testverfahrens zur Prognose eines sekundären Therapieversagens aufgrund einer Hyperreaktivität bei einer Therapie mit Biologicals“. (PI: U. Jappe) (2015-2018)
- ZIM-KF2784701AJ0:,Fördermodul Kooperationsprojekte: „Entwicklung von Testsystemen zum Nachweis von spezifischen IgE-Antikörpern gegen therapeutische monoklonale Antikörper“ (PI: U. Jappe) (2011-2015)
Bundesministerium für Ernährung und Landwirtschaft (BMEL)
- Bundesministerium für Ernährung und Landwirtschaft (BMEL) mit Projektträgerschaft durch die Bundesanstalt für Landwirtschaft und Ernährung (BLE) im Rahmen des Programms zur Innovationsförderung "Entwicklung eines Aptamer-basierten Biosensors zur Detektion und Untersuchung von Allergenen und ihrem allergenen Potential in Lebensmitteln: AptaSens" (PI: U. Jappe) 2020-2023
Stiftungen
- Kanert-Stiftung: Investigation of peanut oilbodies for allergen transport with subsequent analysis of reconstituted oil bodies as vehicles for therapeutic allergen transport (epicutaneous immunotherapy (EPIT)) for peanut allergy (PI. U. Jappe) (2018-2019)
EU - 7. RP
- FP7 RegPot 2010-5_256756FCUB Er zur wissenschaftlichen Förderung des Westbalkans - Partner der Universität Belgrad
Mitglied des Airway Research Center North und Partner im Deutschen Zentrum für Lungenforschung
Mitglied im Companion Diagnostics Network
Mitglied im Network of Severe Allergic Reactions (NORA)
Gentechnik
- Klonierung und Expression von Allergenen
- E. coli, „Clear coli“
- Pichia pastoris
- Insektenzellen (Sf9-Zellen)
- RNA-Silencing spezifischer Gene
- (real-time) PCR
Allergen-/Proteinreinigung
- Wässrige und organische Extraktionsverfahren
- Chromatographie
- Affinitätschromatographie
- Ionenaustauschchromatographie
- Hydrophobe Interaktionschromatographie
- Reversed phase Chromatographie
- Größenausschluss-Chromatographie
- Präparative Gelelektrophorese
Allergen-/Proteinanalytik
- Elektrophorese
- Elektrofokussierung
- SDS-PAGE
- 2D-Elektrophorese
- Differenz-Gelelektrophorese (DIGE)
- Immunologische Methoden
- Immunoplot
- Dot-Test
- ELISA
- ImmunoCAP
Identifizierung
- N-terminale Sequenzierungen
- Massenspektrometrie
Weitere Methoden
- Design von Peptid-Microarrays
- Epitopmapping
- Proteinengineering
- Analyse von Proteinen nach Simulation des humanen Verdaus
- Deglykosylierung von Proteinen
Zellanalytik / Zellkultur
- Kultivierung von Human-/Säugetierzellen
- Zellanalytik per Durchflusszytometrie (z.B. Basophilenaktivierungstest)
- Zellanalytik per Konfokalmikroskopie
- Fluoreszenzmikroskopie, FRET
- Zellbasierte Assaysysteme zur Messung von Signalwegen
Allergie-Kolloquium Borstel
- 23. Allergie-Kolloquium 2010 - (Details sehen)
- 24. Allergie-Kolloquium 2011 - (Details sehen)
- 25. Allergie-Kolloquium 2012 - (Details sehen)
- 26. Allergie-Kolloquium 2013 - (Details sehen)
- 27. Allergie-Kolloquium 2014 - (Details sehen)
- 28. Allergie-Kolloquium 2015 - (Details sehen)
- 29. Allergie-Kolloquium 2016 - (Details sehen)
- 30. Allergie-Kolloquium 2017 - (Details sehen)
- 31. Allergie-Kolloquium 2018 - (Details sehen)
- 32. Allergie-Kolloquium 2019 - (Details sehen)
- 33. Allergie-Kolloquium 2021 - (Details sehen)
- 34. Allergie-Kolloquium 2022 - (Details sehen)
- 35. Allergie-Kolloquium 2023 - (Details sehen)
- 36. Allergie-Kolloquium 2024 - (Details sehen)
Pressemitteilung zum 36. Allergie-Kolloquium und der Kanert-Preisverleihung - 37. Allergie-Kolloquium 2025 - (Details sehen)
Pressemitteilung zum 37. Allergie-Kolloquium und der Kanert-Preisverleihung - 38. Allergie-Kolloquium 2026 - (Detail ansehen)
Lübecker Allergie-Symposium
- 1. Lübecker Allergiesymposium 2010 - (Details sehen)
- 2. Lübecker Allergiesymposium 2011 - (Details sehen)
- 3. Lübecker Allergiesymposium 2012 - (Details sehen)
- 4. Lübecker Allergiesymposium 2013 - (Details sehen)
- 5. Lübecker Allergiesymposium 2015 - (Details sehen)
Internationaler Kongress
7th International Symposium on Molecular Allergology (ISMA), Luxembourg 2017 (Details sehen)
Internationaler Workshop Molekulare Allergologie
WHO/IUIS Allergen Nomenklatur Subkommittee gemeinsam mit der EAACI Task Force BACALL, Borstel 2023
DGAKI-Symposium
Allergieakademie der DGAKI 2019 (Details sehen)
44. Tagung der Norddeutschen Immunologen
NDI3 - New Developments in Immunology, Inflammation and Infection, 2021 (Details ansehen)
2006: Förder-Preis 2006 der Deutschen Kontaktallergie-Gruppe (DKG) (Uta Jappe und Ko-Autoren)
2006: 1. Wissenschaftspreis der Deutschen Gesellschaft für Ultraschall in der Medizin (DGUM) (Uta Jappe und Ko-Autoren)
2007: Kanert-Preis für Allergieforschung (Wolf-Meinhard Becker und Arnd Petersen)
2010: Poster Prize of the European Academy of Allergy and Clinical Immunology (EAACI) (Sandra Rennert, Arnd Petersen, Uta Jappe and co-authors), EAACI Congress in London, England
2011: Preis für den besten Vortrag „Best of Mainz“, Mainzer Allergie-Workshop (Uta Jappe)
2011: Preis für die beste Masterarbeit des FZB 2011: Titel: “Lupine als Auslöser von Nahrungsmittelallergien: Allergen-Identifikation und –Charakterisierung” (Saskia Hellmig)
2012: Preis für das beste Poster, XX. Tagung der Norddeutschen Dermatologischen Gesellschaft, August 2012 in Berlin (Uta Jappe und Ko-Autoren)
2012: Preis für den besten Vortrag; European Academy of Allergy and Clinical Immunology (EAACI) Congress in Genf, Schweiz (Uta Jappe)
2013: Preis für den besten Vortrag; European Academy of Allergy and Clinical Immunology (EAACI) and the World Allergy Organization (WAO) (Uta Jappe), EAACI/WAO Congress in Mailand, Italien
2015: Preis für die beste Masterarbeit des FZB 2015; Titel: “Entwicklung und Validierung eines ELISA-Testsystems zur Quantifizierung des Erdnussallergens Ara h 2 in Muttermilch” (Alexandra Scharf)
2015: Preis für den besten Vortrag, 38. Symposium der Norddeutschen Immunologen, 30.10.2015 in Borstel (Christian Schwager)
2016: Travel Grant and Biolegend Best Talk Award, X. World Immune Regulation Meeting (WIRM), Davos, Switzerland, (Arne Homann)
2016: Preis für den besten Beitrag (Vortrag und Poster) des Retreats des Exzellenz-Clusters Inflammation at Interfaces 2016 (Arne Homann)
2016: Preis für die beste Masterarbeit des FZB 2016: Titel: "Isolierung und Charakterisierung neuer Lupinenallergene" (Arabella Karstedt)
2017: Nachwuchsförderpreis der Deutschen Gemeinschaft für Allergologie und klinische Immunologie (DGAKI) für die beste Publikation (Christian Schwager für die Autoren)
2018: Kanert-Preis für Allergieforschung 2018 (Uta Jappe and Christian Schwager)
2018: Preis für die beste Promotion des FZB (Promotionspreis des Kreises Segeberg) für Christian Schwager; Titel der Dissertation: „Erdnuss-Oleosine: Isolierung, Charakterisierung und Ermittlung der klinischen Relevanz“
2020: Preis für die beste Masterarbeit des FZB 2020: Titel: "Lipophile Erdnussallergene in der Allergiediagnostik" (Vivian Valeska Lelleck)
2022: 2. Mainzer Abstract-Preis der DGAKI für das Kooperationsprojekt zwischen FZB und den Max-Rubner-Instituten Kiel und Karlsruhe an die Autoren Roggensack T, Rodriguez Gomez M, Hui-Zhi-Low, Kappel K, Uta Jappe, Clawin-Rädecker I.
2022: Preis für die beste Masterarbeit des FZB 2022: Titel: "Optimization and validation of the heterologous expression of a food allergen belonging to the group of lipid transfer proteins" (Elisabeth Marie Albert)
2023: Preis für den besten Vortrag (Flash Talk) auf dem Hybrid Congress of the European Academy of Allergy and Clinical Immunology (EAACI) in Hamburg (Theresa Walsemann)
2023: Preis für die beste Presentation der Thematic Poster Session auf dem Hybrid Congress of the European Academy of Allergy and Clinical Immunology (EAACI) in Hamburg (Marua Abu Risha)
2023: Preis für die beste Promotion des FZB (Promotionspreis des Kreises Segeberg) für Theresa Walsemann; Titel der Dissertation: "Identification and characterization of house dust mite allergens influencing the allergic phenotype and improvement of allergy diagnosis in clinical routine."
2024: Travel Grant der European Academy of Allergy and Clinical Immunology (EAACI), EAACI Congress in Valencia, Spanien (Marua Abu Risha)
2024: Travel Grant Award to Marua Abu Risha for the Congress of the European Academy of Allergy and Clinical Immunology (EAACI) in Valencia, Spain
2024: Preis für die beste Masterarbeit des FZB: Titel: "Untersuchung der Toll-Like-Rezeptor-Aktivierung von Markerallergenen der Hausstaubmilbe Dermatophagoides pteronyssinus" (Celina Sachs)
Presse
2022: Prof. Dr. Uta Jappekontinuierlich gelistet seit 2017 in Focus-Gesundheit unter Deutschlands Top Medizinern aus 122 Fachbereichen.
2023, 2024, 2025 und 2026: Prof Dr. Uta Jappe gehört zu den ausgezeichneten Spezialisten der STERN Ärzteliste im Bereich Nahrungsmittelunverträglichkeiten.
2025
Jappe, U, Kleine-Tebbe, J & Brans, R 2025, 'Allergene', ALLERGOLOGIE, Nr. 5, S. 699-716. https://doi.org/10.5414/ALP48699
Jappe, U, Kolaly, T, de Vries, MS, Gülsen, A & Homann, A 2025, 'Connecting Diagnostics and Clinical Relevance of the α-Gal Syndrome-Individual Sensitization Patterns of Patients with Suspected α-Gal-Associated Allergy', Nutrients, Jg. 17, Nr. 9. https://doi.org/10.3390/nu17091541
Pascal, M, Chauhan, J, Knol, E, Bianchini, R, Castells, M, De Las Vecillas, L, Hartmann, K, Izquierdo, E, Jappe, U, Jimenez-Rodriguez, T-W, Levi-Schaffer, F, Mayorga, C, Poli, A, Redegeld, F, Santos, AF, Jensen-Jarolim, E, Bergmann, C, Karagiannis, SN & Bax, HJ 2025, 'Basophil Activation Test: Bridging Allergy and Oncology for Diagnostic, Therapeutic and Prognostic Applications in AllergoOncology: An EAACI Position Paper', ALLERGY. https://doi.org/10.1111/all.16607
Radauer, C, Gadermaier, G, Goodman, RE, Jacquet, A, Jappe, U, Lopata, AL, Pomés, A, Raulf, M, Santos, KS, Zakzuk, J, Zhang, Y, Vitte, J & WHO/IUIS Allergen Nomenclature Sub‐Committee 2025, 'New Allergens Approved by the WHO/IUIS Allergen Nomenclature Sub-Committee in 2021-2024 and Their Significance for Future Diagnostics, Regulation, and Research. An EAACI Task Force Report', ALLERGY. https://doi.org/10.1111/all.70166
Reese, I, Schäfer, C, Ballmer-Weber, B, Beyer, K, Dölle-Bierke, S, van Dullemen, S, Jappe, U, Schnadt, S & Treudler, R 2025, 'Response to Letter to the Editor from Christian Koeder ', Allergologie select, Jg. 9, S. 18-20. https://doi.org/10.5414/ALX02573E
Treudler, R, Worm, M, Bauer , A, Dickel , H, Heine, G, Jappe, U, Klimek, L, Raulf, M, Wedi, B, Wieczorek, D, Francuzik, W, Jakob , T, Pfaar, O, Ring, F, Ruëff, F, Schnadt, S, Werfel, T, Wurpts, G, Zarnowski, J, Zuberbier , T & Brockow, K 2025, 'Berufsbedingte Anaphylaxie – Positionspapier der Deutschen Gesellschaft für Allergologie und Klinische Immunologie (DGAKI)', ALLERGOLOGIE, Jg. 2025, Nr. 48, S. 416-435. https://doi.org/10.5414/ALX02543
Buchbeitrag / Book contribution
Klimek, L, Vogelberg, C &Werfel, T 2025 Weißbuch Allergie in Deutschland. 5 Aufl.
2024
Abu Risha, M, Reddy, KD, Nemani, SSP, Jakwerth, C, Schmidt-Weber, C, Bahmer, T, Hansen, G, von Mutius, E, Rabe, KF, Dittrich, A-M, Grychtol, R, Maison, N, Schaub, B, Kopp, MV, Brinkmann, F, Meiners, S, Jappe, U, Weckmann, M & ALLIANCE Study Group 2024, 'Epigenetic training of human bronchial epithelium cells by repeated rhinovirus infections', ALLERGY, Jg. 79, Nr. 12, S. 3385-3400. https://doi.org/10.1111/all.16388
Abu Risha, M, Rick, E-M, Plum, M & Jappe, U 2024, 'Legume Allergens Pea, Chickpea, Lentil, Lupine and Beyond', Current Allergy and Asthma Reports, Jg. 24, Nr. 9, S. 527-548. https://doi.org/10.1007/s11882-024-01165-7
Darsow, U, Gelincik, A, Jappe, U, Platts-Mills, TA, Ünal, D & Biedermann, T 2024, 'Algorithms in allergy: An algorithm for alpha-Gal syndrome diagnosis and treatment, 2024 update', ALLERGY, Jg. 79, Nr. 11, S. 3169-3172. https://doi.org/10.1111/all.16291
Jappe, U, Bergmann, K-C, Brinkmann, F, Faihs, V, Gülsen, A, Klimek, L, Renz, H, Seurig, S, Taube, C, Traidl, S, Treudler, R, Wagenmann, M, Werfel, T, Worm, M & Zuberbier, T 2024, 'Biologics in allergology and clinical immunology: Update on therapies for atopic diseases, urticaria, and angioedema and on safety aspects focusing on hypersensitivity reactions', Allergologie select, Jg. 8, S. 365-406. https://doi.org/10.5414/ALX02533E
Pascal, M, Bax, HJ, Bergmann, C, Bianchini, R, Castells, M, Chauhan, J, De Las Vecillas, L, Hartmann, K, Álvarez, EI, Jappe, U, Jimenez-Rodriguez, T-W, Knol, E, Levi-Schaffer, F, Mayorga, C, Poli, A, Redegeld, F, Santos, AF, Jensen-Jarolim, E & Karagiannis, SN 2024, 'Granulocytes and mast cells in AllergoOncology-Bridging allergy to cancer: An EAACI position paper', ALLERGY, Jg. 79, Nr. 9, S. 2319-2345. https://doi.org/10.1111/all.16246
Rick, E-M, Woolnough, K, Richardson, M, Monteiro, W, Craner, M, Bourne, M, Cousins, DJ, Swoboda, I, Wardlaw, AJ & Pashley, CH 2024, 'Identification of allergens from Aspergillus fumigatus-Potential association with lung damage in asthma', ALLERGY, Jg. 79, Nr. 5, S. 1208-1218. https://doi.org/10.1111/all.16032
Treudler, R, Worm, M, Bauer, A, Dickel, H, Heine, G, Jappe, U, Klimek, L, Raulf, M, Wedi, B, Wieczorek, D, Francuzik, W, Jakob, T, Pfaar, O, Ring, J, Rueff, F, Schnadt, S, Werfel, T, Wurpts, G, Zarnowski, J, Zuberbier, T & Brockow, K 2024, 'Occupational anaphylaxis: A Position Paper of the German Society of Allergology and Clinical Immunology (DGAKI)', Allergologie select, Jg. 8, S. 407-424. https://doi.org/10.5414/ALX02543E
Turner, MC, Radzikowska, U, Ferastraoaru, DE, Pascal, M, Wesseling, P, McCraw, A, Backes, C, Bax, HJ, Bergmann, C, Bianchini, R, Cari, L, de Las Vecillas, L, Izquierdo, E, Lind-Holm Mogensen, F, Michelucci, A, Nazarov, PV, Niclou, SP, Nocentini, G, Ollert, M, Preusser, M, Rohr-Udilova, N, Scafidi, A, Toth, R, Van Hemelrijck, M, Weller, M, Jappe, U, Escribese, MM, Jensen-Jarolim, E, Karagiannis, SN & Poli, A 2024, 'AllergoOncology: Biomarkers and refined classification for research in the allergy and glioma nexus-A joint EAACI-EANO position paper', ALLERGY, Jg. 79, Nr. 6, S. 1419-1439. https://doi.org/10.1111/all.15994
Buchbeitrag / Book contribution
Dramburg S, Hilger C, Santos AF, de Las Vecillas L, Aalberse RC, Acevedo N, Aglas L, Altmann F, Arruda KL, Asero R, Ballmer-Weber B, Barber D, Beyer K, Biedermann T, Bilo MB, Blank S, Bosshard PP, Breiteneder H, Brough HA, Bublin M, Campbell D, Caraballo L, Caubet JC, Celi G, Chapman MD, Chruszcz M, Custovic A, Czolk R, Davies J, Douladiris N, Eberlein B, Ebisawa M, Ehlers A, Eigenmann P, Gadermaier G, Giovannini M, Gomez F, Grohman R, Guillet C, Hafner C, Hamilton RG, Hauser M, Hawranek T, Hoffmann HJ, Holzhauser T, Iizuka T, Jacquet A, Jakob T, Janssen-Weets B, Jappe U, Jutel M, Kalic T, Kamath S, Kespohl S, Kleine-Tebbe J, Knol E, Knulst A, Konradsen JR, Korošec P, Kuehn A, Lack G, Le TM, Lopata A, Luengo O, Mäkelä M, Marra AM, Mills C, Morisset M, Muraro A, Nowak-Wegrzyn A, Nugraha R, Ollert M, Palosuo K, Pastorello EA, Patil SU, Platts-Mills T, Pomés A, Poncet P, Potapova E, Poulsen LK, Radauer C, Radulovic S, Raulf M, Rougé P, Sastre J, Sato S, Scala E, Schmid JM, Schmid-Grendelmeier P, Schrama D, Sénéchal H, Traidl-Hoffmann C, Valverde-Monge M, van Hage M, van Ree R, Verhoeckx K, Vieths S, Wickman M, Zakzuk J, Matricardi PM, Hoffmann-Sommergruber K. Molecular Allergology Pocket Guide. European Academy of Allergy and Clinical Immunology & John Wiley and sons Ltd. 2024
2023
Albert E, Walsemann T, Behrends J, Jappe U. Lipid transfer protein syndrome in a Northern European patient: An unusual case report. Front Med (Lausanne). 2023 Feb 7;10:1049477. doi: 10.3389/fmed.2023.1049477. eCollection 2023.
Dölle-Bierke S, Höfer V, Francuzik W, Näher F, Bilo M.B., Cichocka-Jarosz E, Felipe Ensina L, Fernandez-Rivas M, García Figueroa B, Hartmann K, Jappe U, Köhli A, Lange L, Maris I, Mustakov T, Nemat K, Ott H, Papadopoulos NG, Pföhler C, Ruëff F, Spindler T, Stock P, Treudler R, Vogelberg C, Wagner N, Worm M. Food-induced anaphylaxis: Data from the European Anaphylaxis Registry. J Allergy Clin Immunol Pract. 2023 Mar 27:S2213-2198(23)00312-4. doi: 10.1016/j.jaip.2023.03.026. Online ahead of print.
Dramburg S, Hilger C, Santos AF, de las Vecillas L, Aalberse RC, Acevedo N, Aglas L, Altmann F, Arruda KL, Asero R, Ballmer-Weber B, Barber D, Beyer K, Biedermann T, Bilo MB, Blank S, Bosshard PP, Breiteneder H, Brough HA, Bublin M, Campbell D, Caraballo L, Caubet JC, Celi G, Chapman MD, Chruszcz M, Custovic A, Czolk R, Davies J, Douladiris N, Eberlein B, Ebisawa M, Ehlers A, Eigenmann P, Gadermaier G, Giovannini M, Gomez F, Grohman R, Guillet C, Hafner C, Hamilton RG, Hauser M, Hawranek T, Hoffmann HJ, Holzhauser T, Iizuka T, Jacquet A, Jakob T, Janssen-Weets B, Jappe U, Jutel M, Kalic T, Kamath S, Kespohl S, Kleine-Tebbe J, Knol E, Knulst A, Konradsen JR, Korošec P, Kuehn A, Lack G, Thuy-My Le, Lopata A, Luengo O, Mäkelä M, Marra AM, Mills C, Morisset M, Muraro A, Nowak-Wegrzyn A, Nugraha R, Ollert M, Palosuo K, Pastorello EA, Patil SU, Platts-Mills TA, Pomés A, Poncet P, Potapova E, Poulsen LK, Radauer C, Radulovic S, Raulf M, Rougé P, Sastre J, Sato S, Scala E, Schmid JM, Schmid-Grendelmeier P, Schrama D, Sénéchal H, Traidl-Hoffmann C, Valverde-Monge M, van Hage M, van Ree R, Verhoeckx K, Vieths S, Wickman M, Zakzuk J, Matricardi PM, Hoffmann-Sommergruber K. EAACI Molecular Allergology User's Guide 2.0. First published: 26 April 2023 https://doi.org/10.1111/pai.13854
Dühring, L, Petry, J, Lilienthal, G-M, Bartsch, YC, Kubiak, M, Pfeufer, C, Lehrian, S, Buhre, JS, Lunding, HB, Kern, C, Behrends, J, Walsemann, T, Gädert, L, Sommer, C, Krüger, L, Blanchard, V, Dehmel, S, Jappe, U, Rahmöller, J & Ehlers, M 2023, 'Sialylation of IgE reduces FcεRIα interaction and mast cell and basophil activation in vitro and increases IgE half-life in vivo', ALLERGY, Jg. 78, Nr. 8, S. 2301-2305. https://doi.org/10.1111/all.15665
Đurašinović, T, Lopandić, Z, Protić-Rosić, I, Nešić, A, Trbojević-Ivić, J, Jappe, U & Gavrović-Jankulović, M 2024, 'Identification of S-adenosyl-l-homocysteine hydrolase from banana fruit as a novel plant panallergen', Food chemistry, Jg. 437, Nr. Pt 1, S. 137782. https://doi.org/10.1016/j.foodchem.2023.137782
Heßler N, Kordowski A, Sasse J, Ahlemann G, Schulz F, Schröder T, Exner A, Jablonski L, Jappe U, Bischoff SC, Grzegorzek M, König IR, Sina C. Study protocol to investigate the efficacy of confocal laser endomicroscopy-based selective single-elimination diet over standard fivefold elimination diet in patients with endomicroscopically proven food intolerance: app-assisted, monocentric, double-blind, randomised and controlled trial in Germany.BMJ Open. 2023 Nov 2;13(11):e072024. doi: 10.1136/bmjopen-2023-072024.
Jappe U. Vegan diet - alternative protein sources as potential allergy risk. Allergo J Int 2023; 32:251-257 doi.org/10.1007/s40629-023-00248-7
Jappe, U. Das Alpha-Gal-Syndrom – eine komplexe Allergie im interdisziplinären Kontext. Aktuelle Ernährungsmedizin 2023; 48(03): 195-208. DOI: 10.1055/a-1870-7328
Plum M, Tjerrild L, Raiber T, Bantleon F, Bantleon S, Miehe M, Jabs F, Seismann H, Möbs C, Pfützner W, Jakob T, Andersen GR, Spillner E. Structural and functional analyses of antibodies specific for modified core N-glycans suggest a role in TH 2 responses. Allergy. 2023;78(1):121-130. doi: 10.1111/all.15417.
Vegan diets from an allergy point of view - Position paper of the DGAKI working group on food allergy. Allergol Select. 2023 Mar 31;7:57-83. doi: 10.5414/ALX02400E. eCollection 2023
2022
Jappe, U 2022, Oral Allergy Syndrome. in E Schmidt (Hrsg.), Diseases of the Oral Mucosa. Springer International Publishing Switzerland, S. 427-434. https://doi.org/10.1007/978-3-030-82804-2_40
Kasper, B, Yue, X, Goldmann, T, Gülsen, A, Kugler, C, Yu, X & Petersen, F 2022, 'Air exposure and cell differentiation are essential for investigation of SARS-CoV-2 entry genes in human primary airway epithelial cells in vitro', Frontiers in Medicine, Jg. 9, S. 897695. https://doi.org/10.3389/fmed.2022.897695
Kleine-Tebbe, J, Brans, R & Jappe, U 2022, 'Allergene - Auslöser der verschiedenen Allergievarianten', Allergo Journal, Jg. 31, Nr. 2, S. 16-31. https://doi.org/10.1007/s15007-022-4980-4
Plum, M, Tjerrild, L, Raiber, T, Bantleon, F, Bantleon, S, Miehe, M, Jabs, F, Seismann, H, Möbs, C, Pfützner, W, Jakob, T, Andersen, GR & Spillner, E 2023, 'Structural and functional analyses of antibodies specific for modified core N-glycans suggest a role in TH 2 responses', ALLERGY, Jg. 78, Nr. 1, S. 121-130. https://doi.org/10.1111/all.15417
Schocker F, Jappe U. Breastfeeding: Maternally transferred allergens in breast milk: Protective or sensitizing? Mol Nutr Food Res. 2022 May 26:e2200066. doi: 10.1002/mnfr.202200066. https://onlinelibrary.wiley.com/doi/10.1002/mnfr.202200066
Walsemann, T, Böttger, M, Traidl, S, Schwager, C, Gülsen, A, Freimooser, S, Rösner, LM, Werfel, T & Jappe, U 2023, 'Specific IgE against the house dust mite allergens Der p 5, 20 and 21 influences the phenotype and severity of atopic diseases', ALLERGY, Jg. 78, Nr. 3, S. 731-742. https://doi.org/10.1111/all.15553
Buchbeitrag (Originalartikel 2021) / Book contribution (original article 2021)
Jappe U, Karstedt A, Warneke D, Hellmig S, Böttger M, Riffelmann FW, Treudler R, Lange L, Abraham S, Dölle-Bierke S, Worm M, Wagner N, Rueff F, Reese G, Knulst AC, Becker WM. Identification and Purification of Novel Low-Molecular-Weight Lupine Allergens as Components for Personalized Diagnostics. In: Food Allergies in Modern Life; eds: Sara Manti, Gian Luigi Marseglia and Salvatore Leonardi. MDPI 2022: 63-83 (www.mdpi.com/journal/nutrients)
2021
Behrends, J, Schwager, C, Hein, M, Scholzen, T, Kull, S & Jappe, U 2021, 'Innovative robust basophil activation test using a novel gating strategy reliably diagnosing allergy with full automation', ALLERGY, Jg. 76, Nr. 12, S. 3776-3788. https://doi.org/10.1111/all.14900
Gülsen, A, König, IR, Jappe, U & Drömann, D 2021, 'Effect of comorbid pulmonary disease on the severity of COVID-19: A systematic review and meta-analysis', Respirology (Carlton, Vic.), Jg. 26, Nr. 6, S. 552-565. https://doi.org/10.1111/resp.14049
Gülsen, A, Ruëff, F & Jappe, U 2021, 'Omalizumab ensures compatibility to bee venom immunotherapy (VIT) after VIT-induced anaphylaxis in a patient with systemic mastocytosis', Allergologie select, Jg. 5, S. 128-132. https://doi.org/10.5414/ALX02196E
Jappe, U, Beckert, H, Bergmann, K-C, Gülsen, A, Klimek, L, Philipp, S, Pickert, J, Rauber-Ellinghaus, MM, Renz, H, Taube, C, Treudler, R, Wagenmann, M, Werfel, T, Worm, M & Zuberbier , T 2021, 'Biologika bei atopischen Erkrankungen: Indikationsstellung, Nebenwirkungsmanagement und neue Entwicklungen', ALLERGOLOGIE, S. 54-80. https://doi.org/10.5414/ALX02197
Jappe, U, Beckert, H, Bergmann, K-C, Gülsen, A, Klimek, L, Philipp, S, Pickert, J, Rauber-Ellinghaus, MM, Renz, H, Taube, C, Treudler, R, Wagenmann, M, Werfel, T, Worm, M & Zuberbier, T 2021, 'Biologics for atopic diseases:Indication, side effect management, and new developments', Allergologie select, Jg. 5, S. 1-25. https://doi.org/10.5414/ALX02197E
Jappe, U, Karstedt, A, Warneke , D, Hellmig, S, Böttger, M, Riffelmann , FW, Treudler, R, Lange, L, Abraham, S, Dölle-Bierke, S, Worm, M, Wagner, N, Ruëff, F, Reese, G, Knulst, AC & Becker, W-M 2021, 'Identification and Purification of Novel Low-Molecular-Weight Lupine Allergens as Components for Personalized Diagnostics', Nutrients, Jg. 13, Nr. 2. https://doi.org/10.3390/nu13020409
Jappe, U, Schwager, C & Gadermaier, G 2021, 'Neue Allergene in der Erdnuss als Ursache einer Anaphylaxie', ALLERGOLOGIE, Jg. 44, Nr. 4, S. 271-276. https://doi.org/10.5414/ALX02179
Kraft, M, Dölle-Bierke, S, Renaudin, J-M, Ruëff, F, Hofmeier, KS, Treudler, R, Pföhler, C, Hawranek, T, Poziomkowska-Gęsicka, I, Jappe, U, Christoff, G, Müller, S, Fernandez-Rivas, M, García, BE, De Vicente Jiménez, TM, Cardona, V, Kleinheinz, A, Kreft, B, Bauer, A, Wagner, N, Wedi, B, Wenzel, M, Bilò, MB & Worm, M 2021, 'Wheat anaphylaxis in adults differs from reactions to other types of food', Journal of Allergy and Clinical Immunology in Clinical Practice, Jg. 9, Nr. 7, S. 2844-2852.e5. https://doi.org/10.1016/j.jaip.2021.03.037
Platts-Mills, TA, Hilger, C, Jappe, U, van Hage, M, Gadermaier, G, Spillner, E, Lidholm, J, Keshavarz, B, Aalberse, RC, van Ree, R, Goodman, RE & Pomés, A 2021, 'Carbohydrate Epitopes Currently Recognized as Targets for IgE Antibodies', ALLERGY, Jg. 76, Nr. 8, S. 2383-2394. https://doi.org/10.1111/all.14802
Recke, A, Becker, W-M & Jappe, U 2021, 'Neu erworbene Milchallergie im Erwachsenenalter', ALLERGOLOGIE, Jg. 44, Nr. 8, S. 592-595.
Schocker, F, Fehrenbach, H & Schromm, AB 2021, 'Mission impossible? A cultural change to support scientific integrity', EMBO REPORTS , Jg. 22, Nr. 7, S. e52334. https://doi.org/10.15252/embr.202052334
Worm, M, Reese, I, Ballmer-Weber, B, Beyer, K, Bischoff, SC, Bohle, B, Brockow, K, Claßen, M, Fischer, PJ, Hamelmann, E, Jappe, U, Kleine-Tebbe, J, Klimek, L, Koletzko, B, Lange, L, Lau, S, Lepp, U, Mahler, V, Nemat, K, Raithel, M, Saloga, J, Schäfer, C, Schnadt, S, Schreiber, J, Szépfalusi, Z, Treudler, R, Wagenmann, M, Werfel, T & Zuberbier, T 2021, 'Update of the S2k guideline on the management of IgE-mediated food allergies', Allergologie select, Jg. 5, S. 195-243. https://doi.org/10.5414/ALX02257E
2020
Dölle-Bierke, S, Plank-Habibi, S, Schäfer , C, Ballmer-Weber, B, Beyer , K, Blümchen, K, Huttegger , I, Jappe, U, Kleine-Tebbe, J, Lange , L, Lau, S, Lepp, U, Mahler , V, Müller, S, Saloga, J, Schnaddt, S, Szépfalusi, Z, Treudler, R, Wassmann-Otto, A, Werfel, T, Zuberbier, T & Reese , I 2020, 'Diätische Implikationen bei ASS-Unverträglickeiten: Stellungnahme der AG Nahrungsmittelallergie der Deutschen Gesellschaft für Allergologie und klinische Immunologie (DGAKI)', Allergo Journal International.
Ferastraoaru, D, Bax, HJ, Bergmann, C, Capron, M, Castells, M, Dombrowicz, D, Fiebiger, E, Gould, HJ, Hartmann, K, Jappe, U, Jordakieva, G, Josephs, DH, Levi-Schaffer, F, Mahler, V, Poli, A, Rosenstreich, D, Roth-Walter, F, Shamji, M, Steveling-Klein, EH, Turner, MC, Untersmayr, E, Karagiannis, SN & Jensen-Jarolim, E 2020, 'AllergoOncology: ultra-low IgE, a potential novel biomarker in cancer-a Position Paper of the European Academy of Allergy and Clinical Immunology (EAACI)', Clinical and Translational Allergy, Jg. 10. https://doi.org/10.1186/s13601-020-00335-w
Gülsen, A, Wedi, B & Jappe, U 2020, 'Hypersensitivity reactions to biologics (part I) allergy as an important differential diagnosis in complex immune-derived adverse events', Allergo Journal, Jg. 29, Nr. 4, S. 32-61. https://doi.org/10.1007/s15007-020-2550-1
Gülsen, A, Wedi, B & Jappe, U 2020, 'Hypersensitivity reactions to Biologics (Part II): classifications and current diagnostic and treatment approaches', Allergo Journal International, Jg. 29, S. 139-154. https://doi.org/10.1007/s40629-020-00127-5
Klimek, L, Pfaar, O, Worm, M, Eiwegger, T, Hagemann, J, Ollert, M, Untersmayr, E, Hoffmann-Sommergruber, K, Vultaggio, A, Agache, I, Bavbek, S, Bossios, A, Casper, I, Chan, S, Chatzipetrou, A, Vogelberg, C, Firinu, D, Kauppi, P, Kolios, A, Kothari, A, Matucci, A, Palomares, O, Szépfalusi, Z, Pohl, W, Hötzenecker, W, Rosenkranz, A, Bergmann, K-C, Bieber, T, Buhl, R, Buters, J, Darsow, U, Keil, T, Kleine-Tebbe, J, Lau, S, Maurer, M, Merk, H, Mösges, R, Saloga, J, Staubach, P, Jappe, U, Rabe, C, Rabe, U, Vogelmeier, C, Biedermann, T, Jung, K, Schlenter, W, Ring, J, Chaker, A, Wehrmann, W, Becker, S, Freudelsperger, L, Mülleneisen, N, Nemat, K, Czech, W, Wrede, H, Brehler, R, Fuchs, T, Tomazic, P-V, Aberer, W, Fink Wagner, A, Horak, F, Wöhrl, S, Niederberger-Leppin, V, Pali-Schöll, I, Roller-Wirnsberger, R, Spranger, O, Valenta, R, Akdis, M, Matricardi, PM, Spertini, F, Khaltaev, N, Michel, J-P, Nicod, L, Schmid-Grendelmeier, P, Idzko, M, Hamelmann, E, Jakob, T, Werfel, T, Wagenmann, M, Taube, C, Jensen-Jarolim, E, Korn, S, Hentges, F, Schwarze, J, O Mahony, L, Knol, E, Del Giacco, S, Chivato, T, Bousquet, J, Zuberbier, T, Akdis, C & Jutel, M 2020, 'Anwendung von Biologika bei allergischen und Typ-2-entzündlichen Erkrankungen in der aktuellen Covid-19-Pandemie: Positionspapier des Ärzteverbands Deutscher Allergologen (AeDA)A, der Deutschen Gesellschaft für Allergologie und klinische Immunologie (DGAKI)B, der Gesellschaft für Pädiatrische Allergologie und Umweltmedizin (GPA)C, der Österreichischen Gesellschaft für Allergologie und Immunologie (ÖGAI)D, der Luxemburgischen Gesellschaft für Allergologie und Immunologie (LGAI)E, der Österreichischen Gesellschaft für Pneumologie (ÖGP)F in Kooperation mit der deutschen, österreichischen, und schweizerischen ARIA-GruppeG und der Europäischen Akademie für Allergologie und Klinische Immunologie (EAACI)H', Allergo Journal, Jg. 29, Nr. 4, S. 14-27. https://doi.org/10.1007/s15007-020-2553-y
Klimek, L, Pfaar, O, Worm, M, Eiwegger, T, Hagemann, J, Ollert, M, Untersmayr, E, Hoffmann-Sommergruber, K, Vultaggio, A, Agache, I, Bavbek, S, Bossios, A, Casper, I, Chan, S, Chatzipetrou, A, Vogelberg, C, Firinu, D, Kauppi, P, Kolios, A, Kothari, A, Matucci, A, Palomares, O, Szépfalusi, Z, Pohl, W, Hötzenecker, W, Rosenkranz, AR, Bergmann, K-C, Bieber, T, Buhl, R, Buters, J, Darsow, U, Keil, T, Kleine-Tebbe, J, Lau, S, Maurer, M, Merk, H, Mösges, R, Saloga, J, Staubach, P, Jappe, U, Rabe, KF, Rabe, U, Vogelmeier, C, Biedermann, T, Jung, K, Schlenter, W, Ring, J, Chaker, A, Wehrmann, W, Becker, S, Freudelsperger, L, Mülleneisen, N, Nemat, K, Czech, W, Wrede, H, Brehler, R, Fuchs, T, Tomazic, P-V, Aberer, W, Fink-Wagner, A-H, Horak, F, Wöhrl, S, Niederberger-Leppin, V, Pali-Schöll, I, Pohl, W, Roller-Wirnsberger, R, Spranger, O, Valenta, R, Akdis, M, Matricardi, PM, Spertini, F, Khaltaev, N, Michel, J-P, Nicod, L, Schmid-Grendelmeier, P, Idzko, M, Hamelmann, E, Jakob, T, Werfel, T, Wagenmann, M, Taube, C, Jensen-Jarolim, E, Korn, S, Hentges, F, Schwarze, J, O Mahony, L, Knol, EF, Del Giacco, S, Chivato Pérez, T, Bousquet, J, Bedbrook, A, Zuberbier, T, Akdis, C & Jutel, M 2020, 'Use of biologicals in allergic and type-2 inflammatory diseases during the current COVID-19 pandemic: Position paper of Ärzteverband Deutscher Allergologen (AeDA)A, Deutsche Gesellschaft für Allergologie und Klinische Immunologie (DGAKI)B, Gesellschaft für Pädiatrische Allergologie und Umweltmedizin (GPA)C, Österreichische Gesellschaft für Allergologie und Immunologie (ÖGAI)D, Luxemburgische Gesellschaft für Allergologie und Immunologie (LGAI)E, Österreichische Gesellschaft für Pneumologie (ÖGP)F in co-operation with the German, Austrian, and Swiss ARIA groupsG, and the European Academy of Allergy and Clinical Immunology (EAACI)H', Allergologie select, Jg. 4, S. 53-68. https://doi.org/10.5414/ALX02166E
Kraft, M, Hofmeier, KS, Ruëff, F, Pföhler, C, Renaudin, J-M, Bilò, MB, Treudler, R, Lang, R, Cichoka-Jarosz, E, Fernandez-Rivas, M, Christoff, G, Papadopoulus, NG, Ensina, LF, O'B Hourihane, J, Maris, I, Köhli, A, García, BE, Jappe, U, Vogelberg, C, Ott, H, Lange, L, Spindler, T, Dölle-Bierke, S & Worm, M 2020, 'Risk factors and characteristics of biphasic anaphylaxis', Journal of Allergy and Clinical Immunology in Clinical Practice, Jg. 8, Nr. 10, S. 3388-3395.e6. https://doi.org/10.1016/j.jaip.2020.07.036
Krause, T, Röckendorf, N, Frey, B, Sinnecker, H, Schwager, C, Möckel, S, Jappe, U & Frey, A 2020, 'IgE Epitope Profiling for Allergy Diagnosis and Therapy – Parallel Analysis of a Multitude of Potential Linear Epitopes Using a High Throughput Screening Platform', FRONTIERS IN IMMUNOLOGY, Jg. 11, Nr. 11, 565243, S. 565243. https://doi.org/10.3389/fimmu.2020.565243, https://doi.org/10.3389/fimmu.2020.565243
Platts-Mills, TAE, Commins, SP, Biedermann, T, van Hage, M, Levin, M, Beck, LA, Diuk-Wasser, M, Jappe, U, Apostolovic, D, Minnicozzi, M, Plaut, M & Wilson, JM 2020, 'On the cause and consequences of IgE to galactose-α-1,3-galactose: A report from the National Institute of Allergy and Infectious Diseases Workshop on Understanding IgE-Mediated Mammalian Meat Allergy', The Journal of allergy and clinical immunology, Jg. 145, Nr. 4, S. 1061-1071. https://doi.org/10.1016/j.jaci.2020.01.047
Ramírez Caballero, L, Kny, C, Treudler, R, Simon, JC, Kern, K, Jappe, U & Szardenings, M 2020, 'Identification of Seasonal Variations of Antibodies against PR-10-Specific Epitopes Can Be Improved Using Peptide-Phage Display', INTERNATIONAL ARCHIVES OF ALLERGY AND IMMUNOLOGY , Jg. 181, Nr. 12, S. 919–925 . https://doi.org/10.1159/000509995
Schocker, F, Kull, S, Schwager, C, Behrends, J & Jappe, U 2020, 'Individual Sensitization Pattern Recognition to Cow’s Milk and Human Milk Differs for Various Clinical Manifestations of Milk Allergy', Nutrients, Jg. 11, Nr. 6, S. 95-105. https://doi.org/10.3390/books978-3-03928-029-2
Wanka, L & Jappe, U 2021, 'Trained Immunity and Allergy: State of the Art and Future Perspectives', ALLERGY, Jg. 76, Nr. 4, S. 1265-1267. https://doi.org/10.1111/all.14617
2019
González Roldán, N, Engel, R, Düpow, S, Jakob, K, Koops, F, Orinska, Z, Vigor, C, Oger, C, Galano, J-M, Durand, T, Jappe, U & Duda, KA 2019, 'Lipid Mediators From Timothy Grass Pollen Contribute to the Effector Phase of Allergy and Prime Dendritic Cells for Glycolipid Presentation', FRONTIERS IN IMMUNOLOGY, Jg. 10, S. 974. https://doi.org/10.3389/fimmu.2019.00974
Gülsen, A, Wallis, S & Jappe, U 2021, 'Combination of immunotherapies for severe allergic asthma', Journal of Asthma, Jg. 58, Nr. 1, S. 75-78. https://doi.org/10.1080/02770903.2019.1658204
Jappe, U, Schwager, C, Schromm, A, Gonzalez Roldan, N, Stein, K, Heine, H & Duda, K 2019, 'Lipophilic allergens, different modes of allergen-lipid interaction and their impact on asthma and allergy', FRONTIERS IN IMMUNOLOGY, Jg. 10, S. 122. https://doi.org/10.3389/fimmu.2019.00122
Recke, A, Massalme, EG, Jappe, U, Steinmüller-Magin , L, Schmidt, J, Hellenbroich, Y, Gillessen-Kaesbach , G, Zillikens, D & Hartmann, K 2019, 'Identification of the recently described plasminogen gene mutation p.Lys330Glu in a family from Northern Germany with hereditary angioedema', Clinical and Translational Allergy, Jg. 9, Nr. 9, S. 9. https://doi.org/10.1186/s13601-019-0247-x
Schocker, F 2019, 'Allergieprophylaxe: Erdnüsse in der Stillzeit?', DEUTSCHE HEBAMMENZEITSCHRIFT, Jg. 71, S. 22-28. https://www.dhz-online.de/no_cache/archiv/archiv-inhalt-heft/archiv-detail-abo/artikel/erdnuesse-in-der-stillzeit/
Schocker, F, Kull, S, Schwager, C, Behrends, J & Jappe, U 2019, 'Individual Sensitization Pattern Recognition to Cow’s Milk and Human Milk Differs for Various Clinical Manifestations of Milk Allergy', Nutrients, Jg. 11, Nr. 6, S. 1331. https://doi.org/10.3390/nu11061331
2018
Anemüller, W, Mohrs, M, Brans, R, Homann, A & Jappe, U 2018, 'Alpha-Gal-assoziierte verzögerte Anaphylaxie gegen rotes Fleisch als Berufskrankheit.', Der Hautarzt; Zeitschrift für Dermatologie, Venerologie, und verwandte Gebiete, Jg. 69, Nr. 10, S. 848-852. https://doi.org/10.1007/s00105-018-4224-4
Gülsen, A & Jappe, U 2019, 'Lipid transfer protein sensitization in an apple-allergic patient: a case report from northern Europe', European Annals of Allergy and Clinical Immunology, Jg. 51, Nr. 2, S. 80-83. https://doi.org/10.23822/EurAnnACI.1764-1489.63
Jappe, U & Breiteneder, H 2019, 'Peanut allergy - Individual molecules as a key to precision medicine', ALLERGY, Jg. 74, Nr. 2, S. 216-219. https://doi.org/10.1111/all.13625
Jappe, U 2018, 'Die Nomenklatur der Allergene: Allergen nomenclature', HAUTARZT, Jg. 69, Nr. 1, S. 90-91. https://link.springer.com/article/10.1007/s00105-017-4103-4
Jappe, U 2018, 'Nahrungsmittelallergie: Theorie und Praxis.', DEUTSCHE MEDIZINISCHE WOCHENSCHRIFT, Jg. 143, S. 713-721. https://www.thieme-connect.com/products/ejournals/abstract/10.1055/s-0043-121014
McIntyre, M 2018, 'Ohrmuschel und Gehörgang: wann ist es eine Allergie?', ALLERGOLOGIE, Jg. 41, Nr. 3, S. 105-110. https://doi.org/10.5414/ALX1983
Nikolic, J, Kull, S, Schocker, F, Nesic, A & Gavrovic-Jankulovic, M 2018, 'Employment of proteomic and immunological based methods for the identification of catalase as novel allergen from banana', Journal of Proteomics, Jg. 175, S. 87-94. https://doi.org/10.1016/j.prot.2018.01.007
Pomés, A, Davies, JM, Gadermaier, G, Hilger, C, Holzhauser, T, Lidholm, J, Lopata, AL, Mueller, GA, Nandy, A, Radauer, C, Chan, SK, Jappe, U, Kleine-Tebbe, J, Thomas, WR, Chapman, MD, van Hage, M, van Ree, R, Vieths, S, Raulf, M, Goodman, RE & WHO IUIS Allergen Nomenclature Sub-Committee 2018, 'WHO/IUIS Allergen Nomenclature: Providing a common language', MOLECULAR IMMUNOLOGY, Jg. 100, S. 3-13. https://doi.org/10.1016/j.molimm.2018.03.003
Schocker, F, Recke, A, Kull, S, Worm, M, Behrends, J & Jappe, U 2018, 'Reply to Chirumbolo et al', Pediatric Allergy and Immunology, Jg. 29, Nr. 4, S. 461-462. https://doi.org/10.1111/pai.12892
Schocker, F, Recke, A, Kull, S, Worm, M & Jappe, U 2018, 'Persistent cow's milk anaphylaxis from early childhood monitored by IgE and BAT to cow's and human milk under therapy', Pediatric Allergy and Immunology, Jg. 29, Nr. 2, S. 210-214. https://doi.org/10.1111/pai.12843
Leitung der Forschungsgruppe

(Anmeldung für Patienten: Tel.: 0451-500-44165; -44195; -44196 sowie unter der E-Mailadresse ambulanz.innere.luebeck@uksh.de)
Wissenschaftliche Mitarbeiter:innen

Technische Mitarbeiter:innen
Alumni
- MSC Elisabeth Albert
- Isabell Behrens
- MSC Blessing Asuquo Esu
- Dr. med. Askin Gülsen
- MSC Saskia Hellmig
- Dr. Arne Homann
- MSc Arabella Karstedt
- Dr. med. Tamina Kolaly
- MSC Miriam Kosakowski
- Dr. Skadi Kull
- MSC Vivian Lelleck
- Dr. Sandra Minge
- MSC Muriel Mrose
- Carolin Murawski
- Dr. med. Annika Opitz
- Laura Paulsen
- Prof. Dr. Arnd Petersen
- Dr. Sandra Rennert
- MSC Celina Sachs
- MSC Alexandra Scharf
- Dr. Frauke Schocker
- Dr. Christian Schwager
- Dr. Mareike de Vries
- Theresa Walsemann
- Dr. Lizzy Wanka